Mantenimiento de la estabilidad genómica en bacterias
Resumen de Investigación:
El mantenimiento de la estabilidad genómica depende en gran medida de la replicación fiel del DNA. Sin embargo, el daño continuo que sufren los genomas por agentes genotóxicos ha hecho necesario la emergencia de mecanismos de reparación que prevengan los efectos deletéreos que la permanencia de dichas lesiones podría causar.
Nuestro principal objetivo es el estudio a nivel molecular de los mecanismos responsables del mantenimiento de la información genética en bacterias, mediante el análisis de las propiedades enzimáticas de proteínas de reparación de bacterias modelo, como la Gram positiva Bacillus subtilis, cuyas células vegetativas y esporas están expuestas a condiciones medioambientales extremas, causantes de múltiples daños en el DNA, así como la Gram negativa Pseudomonas aeruginosa.
En ese sentido. durante los últimos años hemos estudiado las funciones catalíticas de la DNA polimerasa de B. subtilis perteneciente a la familia X (PolXBs). Hemos mostrado que esta polimerasa posee, además de la actividad de polimerización, una actividad 3’-5’ exonucleasa, AP endonucleasa, 3’-fosfatasa y 3’-fosfodiesterasa. Todas estas actividades nucleolíticas comparten el mismo centro activo en el dominio PHP C-terminal, específico del subgrupo de las PolXs bacterianas y de arqueas. En coordinación con la actividad de polimerización, esas actividades nucleolíticas permiten a la enzima reconocer sitios abásicos (AP), hidrolizarlos y posteriormente restaurar (reparar) el nucleótido original no dañado, así como procesar extremos 3’ dañados que pueden surgir tras la exposición del DNA a agentes genotóxicos.
Muchas bacterias están provistas de un sistema de unión de extremos no homólogos (NHEJ), responsable de la reparación de las dobles roturas del DNA (DSBs), el tipo de lesión más tóxico ya que es letal para las células en división si no es reparada. Dicha ruta de reparación está constituida por un homodímero de Ku y una DNA ligasa multicatalítica y dependiente de ATP (LigD). Los estudios bioquímicos previos de las LigDs de bacterias permitieron identificar la presencia de actividades de polimerización, ligasa, y en algunos miembros una actividad fosfoesterasa. Gracias a la puesta a punto de reacciones de reparación por escisión de bases (BER) in vitro, utilizando sustratos de DNA que portan sitios AP y las LigDs purificadas procedentes de B. subtilis y P. aeruginosa, hemos podido identificar la presencia adicional de una nueva actividad 5’-2-deoxyribosa-5-fosfato (dRP) liasa en el dominio ligasa (LigDom). Esta actividad se coordina con las de polimerización y ligasa para permitir la reparación eficiente de DNA que contienen sitios AP. Así pues, las LigD contienen, en la misma cadena polipeptídica las tres actividades requeridas en los últimos pasos de BER, sugiriendo que el papel en la reparación del DNA de estas enzimas no está restringido a la ruta NHEJ sino que potencialmente puede formar parte de otras rutas de reparación.
Los sitios AP son las lesiones genómicas más comunes y frecuentemente se asocian a DSBs. La presencia de dichas lesiones cerca de la rotura puede bloquear el paso de ligación final, por lo que dichas lesiones han de ser escindidas. Nuestros últimos resultados muestran la presencia de una actividad AP-liasa adicional en el dominio de polimerización (PolDom) de la LigD de B. subtilis que procesa sitios AP, específicamente cuando se encuentran próximos a extremos 5’-recesivos, y a través de la formación de un complejo preternario-precatalítico con iones Mn2+ y con el ribonucleótido entrante complementario al nucleótido molde que se encuentra frente al sitio AP, garantizando de esa forma el acoplamiento de la eliminación de dicha lesión con la unión de los extremos de la rotura por la LigD (ver Figura 1).
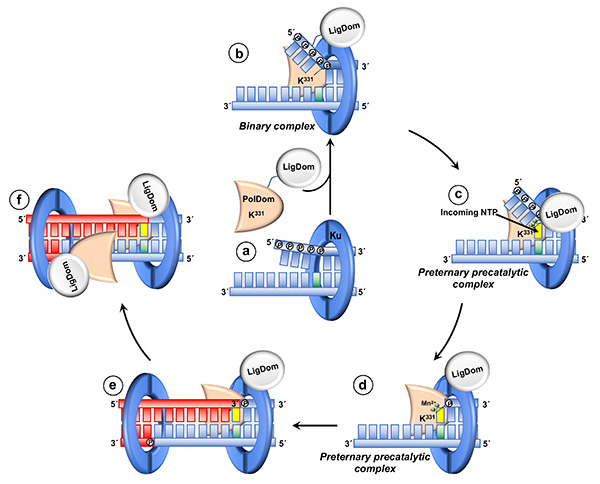
Figura 1. Acoplamiento de la escisión de los sitios AP ala reacción de unión de extremos por la LigD de B. subtillis. Después de producirse la rotura, el extremo del DNA se enhebra a través del anillo interno del homodímero Ku (a). La localización del sitio AP próximo al extremo 5’ podría promover la deshibridación parcial del DNA, haciendo al sitio AP accesible. Una vez reclutada por Ku, LigD formaría un complejo con el DNA, probablemente a través de la interacción entre la Lys331 y uno de los fosfatos del enlace fosfodiéster entre le sitio AP y el siguiente nucleótido en 3’ (b). El nucleótido posicionado frente al sitio AP dirige la unión del ribonucleótido complementario, formando un par de bases Watson-Crick en el sitio de polimerización del PolDom (c). Una vez que el complejo preternario-precatalítico es estabilizado, la proteína incide en el sitio AP, liberando el fragmento 5’ de la cadena, generando un nuevo extremo 5’-P (d). El PolDom media la posterior sinápsis entre los extremos 3’ protuberantes procedentes de las roturas, catalizando la adición in trans del nucleótido al extremo 3’ del primer (e). Finalmente, el LigDom sella ambos extremos (f).

| Apellidos | Nombre | Laboratorio | Ext.* | Categoría profesional | |
|---|---|---|---|---|---|
| Alba Fernández | Lucía | 409 | 4463 | lucia.alba(at)cbm.csic.es | M1 |
| Díaz Arco | Silvia | 409 | 4463 | silvia.diaz(at)cbm.csic.es | M3 Predoc.formación |
| Prado Díaz | Alicia del | 409 | 4463 | adelprado(at)cbm.csic.es | E. Técnicos Especializados de Organismos Públicos de Investigación |
| Vega José | Miguel de | 409 | 4717 | mdevega(at)cbm.csic.es | E. Investigadores Científicos de Organismos Públicos |
Publicaciones relevantes:
- Rodríguez, G., Martín, M.T. and de Vega, M. (2019) An array of basic residues is essential for the nucleolytic activity of the PHP domain of bacterial/archaeal PolX DNA polymerases. Sci. Rep. 9:9947
- de Ory, A., Carabaña, C., and de Vega, M. (2019) Bacterial Ligase D preternary-precatalytic complex performs efficient abasic sites processing at double strand breaks during nonhomologous end joining. Nucleic Acids Res. 47(10), 5276-5292.
- Fernández-García, J.L., de Ory, A., Brussaard, C.P.D. and de Vega, M. (2017) Phaeocystis globosa virus DNA polymerase: a "Swiss Army knife", multifunctional DNA polymerase-lyase-ligase for base excision repair. Sci. Rep. 7:6907
- Zafra, O., Pérez de Ayala, L. and de Vega, M. (2017) The anti/syn conformation of 8´-oxo-7,8-dihydro-2´-deoxyguanosine is modulated by Bacillus subtilis PolX active site residues His255 and Asn263. Efficient processing of damaged 3´-ends. DNA Repair 52: 59-69
- de Ory, A., Nagler, K., Carrasco, B., Raguse, M., Zafra, O., Moeller, R. and de Vega, M. (2016) Identification of a conserved 5´-dRP lyase activity in bacterial DNA repair ligase D and its potential role in base excision repair. Nucleic Acids Res. 44(4): 1833-44
- de Ory, A., Zafra, O. and de Vega, M. (2014) Efficient processing of abasic sites by bacterial nonhomologous end-joining ku proteins. Nucleic Acids Res. 42(21): 13082-95.
- de Vega M. (2013) The minimal Bacillus subtilis nonhomologous end joining repair machinery. PLoS ONE 8(5): e64232.
- Baños,. B, Villar, L., Salas, M., and de Vega M. (2012) DNA stabilization at the Bacillus subtilis PolX core: a binding model to coordinate polymerase, AP-endonuclease and 3'-5' exonuclease activities. Nucleic Acids Res. 40(19):9750-62.
- Baños, B., Villar, L. Salas, M. and de Vega, M. (2010) Intrinsic apurinic/apyrimidinic (AP) endonuclease activity enables Bacillus subtilis DNA polymerase X to recognize, incise, and further repair abasic sites. Proc. Nat. Acad. Sci. USA 107(45): 19219-19224.
- Baños, B., Lázaro, J.M., Villar, L., Salas, M. and de Vega, M. (2008) Editing of misaligned 3’-termini by an intrinsic 3’-5’ exonuclease activity residing in the PHP domain of a family X DNA polymerase. Nucleic Acids Res. 36(18): 5736-5749
Tesis doctorales:
- Irene Rodríguez García (2006). La DNA polimerasa del bacteriófago ø29: Análisis mutacional de la interacción con la proteína terminal. Base estructural de la procesividad y la capacidad de desplazamiento de banda. Universidad Autónoma de Madrid. Directores: Miguel de Vega & Margarita Salas
- Patricia Pérez Arnaiz (2008). Relación estructura-función en la DNA polimerasa del bacteriófago ø29. Papel del dominio intermedio de la proteína terminal en el reconocimineto específico de la DNA polimerasa. Universidad Autónoma de Madrid. Director: Miguel de Vega
- Elisa Longás Torné (2008). Caracterización funcional de las DNA polimerasas de los bacteriófagos Nf y GA-1. Estudio del mecanismo de iniciación en la replicación con proteína terminal. Universidad Autónoma de Madrid. Directores: Miguel de Vega & Margarita Salas
- Benito Baños Piñero (2011). Caracterización funcional de la DNA polimerasa X de Bacillus subtilis. Universidad Autónoma de Madrid. Director: Miguel de Vega
- Alicia del Prado Díaz (2015) Estudios estructurales y funcionales de la DNA polimerasa y la proteína terminal del bacteriófago ø29. Universidad Autónoma de Madrid. Directores: Miguel de Vega & Margarita Salas
- Ana de Ory López (2016). Análisis bioquímico de las proteínas de reparación del DNA Ku y Ligasa D de Bacillus subtilis. Universidad Autónoma de Madrid. Director: Miguel de Vega
- Mª Eugenia Santos del Río (2017). Papel del motivo LExE de la DNA polimerasas que inician con proteína terminal en la interacción con el nucleótido entrante. Estabilización de los sustratos en el centro activo de polimerización mediada por el subdominio TPR1. Universidad Autónoma de Madrid. Directores: Miguel de Vega & Margarita Salas
Patentes:
- Método para la replicación, amplificación o secuenciación de un ADN molde. Inventors: Margarita Salas Falgueras, Miguel de Vega José, José M. Lázaro Bolos, Luis Blanco Dávila, Mario Mencía Caballero. Owner:CSIC. Priority number: P200930412. Priority date: July 2, 2009. PCT/ES2010/070456 presented July 1, 2010. Licensed to XPol Biotech, S.L.
- Quimera de ADN polimerasa del fago ø29. Inventors: Margarita Salas Falgueras, Miguel de Vega José, José M. Lázaro Bolos, Luis Blanco Dávila, Mario Mencía Caballero. Owner: CSIC. Priority number: P200930413. Priority date: July 2, 2009. PCT/ES2010/070454 presented on July 1, 2010. Licensed to XPol Biotech, S.L.
- Método de amplificación de ADN basado en los orígenes de replicación del bacteriófago phi29 y secuencias nucleotídicas asociadas.Inventors: Margarita Salas Falgueras, Mario Mencía Caballero,Miguel de Vega José, Pablo Gella Montero, José M. Lázaro Bolos. Owner: CSIC. Priority number: P201130288. Priority date: March 3, 2011.




















