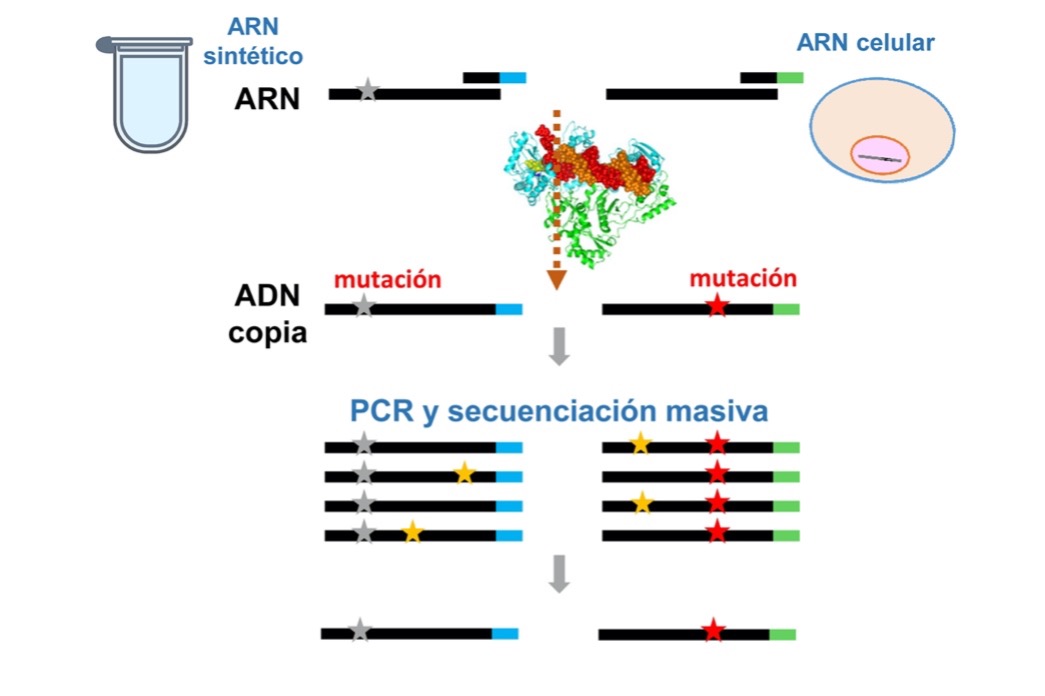
Un equipo de investigadores del Centro de Biología Molecular Severo Ochoa (CBM-CSIC-UAM) ha presentado un método innovador y relativamente sencillo para medir con precisión la tasa de error de la transcriptasa inversa del virus VIH-1. Esta enzima produce ADN a partir de una plantilla de ARN, un proceso clave en la replicación de retrovirus como el VIH. La tasa de error, o fidelidad, de una polimerasa como la transcriptasa inversa indica su capacidad para copiar ácidos nucleicos con precisión, siendo esto crucial para evitar mutaciones que pueden afectar a la estabilidad genética o capacidad replicativa del virus. Este avance, basado en técnicas de secuenciación masiva (Next Generation Sequencing o NGS), ofrece una alternativa más sencilla, económica y precisa que los métodos tradicionales, que suelen ser complejos y laboriosos.
El estudio, recientemente publicado, destaca la implementación de un ensayo de secuenciación masiva, capaz de generar resultados robustos con un número reducido de secuencias. Además, por primera vez, los investigadores utilizaron una secuencia viral como plantilla para estimar las tasas de error de la transcriptasa inversa del VIH-1, evaluando parámetros técnicos fundamentales y utilizando ARN de diferentes orígenes.
Los resultados son reveladores: la fidelidad de la transcriptasa inversa es ocho veces mayor cuando la síntesis de ADN se realiza a concentraciones de magnesio más bajas de las habituales. Esto coincide con estudios anteriores y subraya el impacto de las condiciones experimentales en la precisión de la enzima. Sorprendentemente, los investigadores también encontraron que las tasas de error de la transcriptasa inversa son similares para la misma secuencia de ARN, independientemente de si este ARN se ha obtenido sintéticamente o a partir de células infectadas por el VIH.
Esquema que muestra la generación de mutaciones en el proceso de retrotranscripción y posterior amplificación por PCR, utilizado en los procedimientos de secuenciación masiva / Luis Menéndez Arias & Javier Martínez del Río
Este método ofrece un avance significativo para el estudio de la fidelidad de polimerasas de retrovirus, con implicaciones importantes en transcriptómica, influyendo en la detección de ARNs presentes en poca cantidad, así como para la identificación de modificaciones en el ARN. Además, los resultados obtenidos podrían contribuir al desarrollo de enzimas mejoradas para diversas aplicaciones biotecnológicas.
“Nuestro trabajo no solo aporta un enfoque nuevo, sino que también establece un marco para evaluar la fidelidad de enzimas retrovirales en distintos contextos”, señala Luis Menéndez Arias, autor de correspondencia del estudio y director del grupo de investigación “Retrotranscriptasa del virus de la inmunodeficiencia humana y terapia antirretroviral” en el CBM.
Este avance promete beneficiar tanto a la investigación básica como a aplicaciones biotecnológicas, marcando un paso importante en el desarrollo de herramientas para la mejora de tecnologías basadas en transcriptasas inversas.
Referencia
Martínez Del Río J, Frutos-Beltrán E, Sebastián-Martín A, Lasala F, Yasukawa K, Delgado R, Menéndez-Arias L. HIV-1 Reverse Transcriptase Error Rates and Transcriptional Thresholds Based on Single-strand Consensus Sequencing of Target RNA Derived From In Vitro-transcription and HIV-infected Cells. J Mol Biol. 2024 Nov 15;436(22):168815. DOI: 10.1016/j.jmb.2024.168815
Contacto
CBM Comunicación: comunicacion@cbm.csic.es
Luis Menéndez Arias: lmenendez@cbm.csic.es