Programa Científico
Dinámica y función del genoma
GRUPO DE INVESTIGACIÓN
Mantenimiento de la estabilidad genómica en bacterias

Miguel de Vega José
Para entender cómo se lleva a cabo la transmisión fiel y eficiente de la información genética, estudiamos las propiedades bioquímicas de las DNA polimerasas que capacitan al enzima para replicar y reparar de forma eficiente y precisa el genoma. Para ello, realizamos ensayos bioquímicos con las DNA polimerasas purificadas y analizamos su comportamiento sobre diferentes sustratos de DNA.
Investigación
El mantenimiento de la estabilidad genómica depende en gran medida de la replicación fiel del DNA. Sin embargo, el daño continuo que sufren los genomas por agentes genotóxicos ha hecho necesario la emergencia de mecanismos de reparación que prevengan los efectos deletéreos que la permanencia de dichas lesiones podría causar.
Nuestro principal objetivo es el estudio a nivel molecular de los mecanismos responsables del mantenimiento de la información genética en bacterias, mediante el análisis de las propiedades enzimáticas de proteínas de reparación de bacterias modelo, como la Gram positiva Bacillus subtilis y las Gram negativas Pseudomonas aeruginosa y Escherichia coli.
En ese sentido durante los últimos años hemos estudiado las funciones catalíticas de la DNA polimerasa de B. subtilis perteneciente a la familia X (PolXBs). Hemos demostrado que esta polimerasa posee, además de la actividad de polimerización, una actividad 3’-5’ exonucleasa, AP endonucleasa, 3’-fosfatasa y 3’-fosfodiesterasa. Todas estas actividades nucleolíticas comparten el mismo centro activo en el dominio PHP C-terminal, específico del subgrupo de las PolXs bacterianas y de arqueas. En coordinación con la actividad de polimerización, esas actividades nucleolíticas permiten a la enzima reconocer sitios abásicos (AP), hidrolizarlos y posteriormente restaurar (reparar) el nucleótido original no dañado, así como procesar extremos 3’ dañados que pueden surgir tras la exposición del DNA a agentes genotóxicos.
Muchos miembros bacterianos disponen de un sistema de unión de extremos no homólogos (NHEJ) responsable de reparar las roturas de doble cadena (DSB), las lesiones del ADN más peligrosas, ya que son letales para las células en división si no se reparan a tiempo. Esta vía de reparación está constituida por un homodímero de Ku y una ligasa específica y multifuncional dependiente de ATP (LigD). Análisis bioquímicos previos de la LigD bacteriana permitieron identificar actividades de polimerización, ligasa y fosfoesterasa. Recientemente hemos caracterizado la presencia adicional de una inesperada actividad 5′-2-deoxirribosa-5-fosfato (dRP) liasa en el dominio ligasa (LigDom) de la LigD de B. subtilis y P.aeruginosa. Esta actividad se coordina con las actividades de polimerización y ligasa para permitir la reparación eficaz de un ADN que contiene un sitio AP en una reacción de reparación por escisión de bases (BER) reconstituida in vitro. Por lo tanto, LigD tiene en la misma cadena polipeptídica las tres actividades requeridas en los últimos pasos de BER, lo que sugiere que su papel en la reparación del ADN no se limita a la vía NHEJ, sino que se expande más allá, siendo potencialmente activo en vías de reparación adicionales. Además, hemos identificado una nueva actividad AP-liasa en el PolDom de LigD que escinde sitios AP específicamente cuando están proximales a extremos 5’recesivos y a través de la formación de un complejo precatalítico preterminal con iones Mn2+ y un ribonucleótido entrante complementario al nucleótido de templado
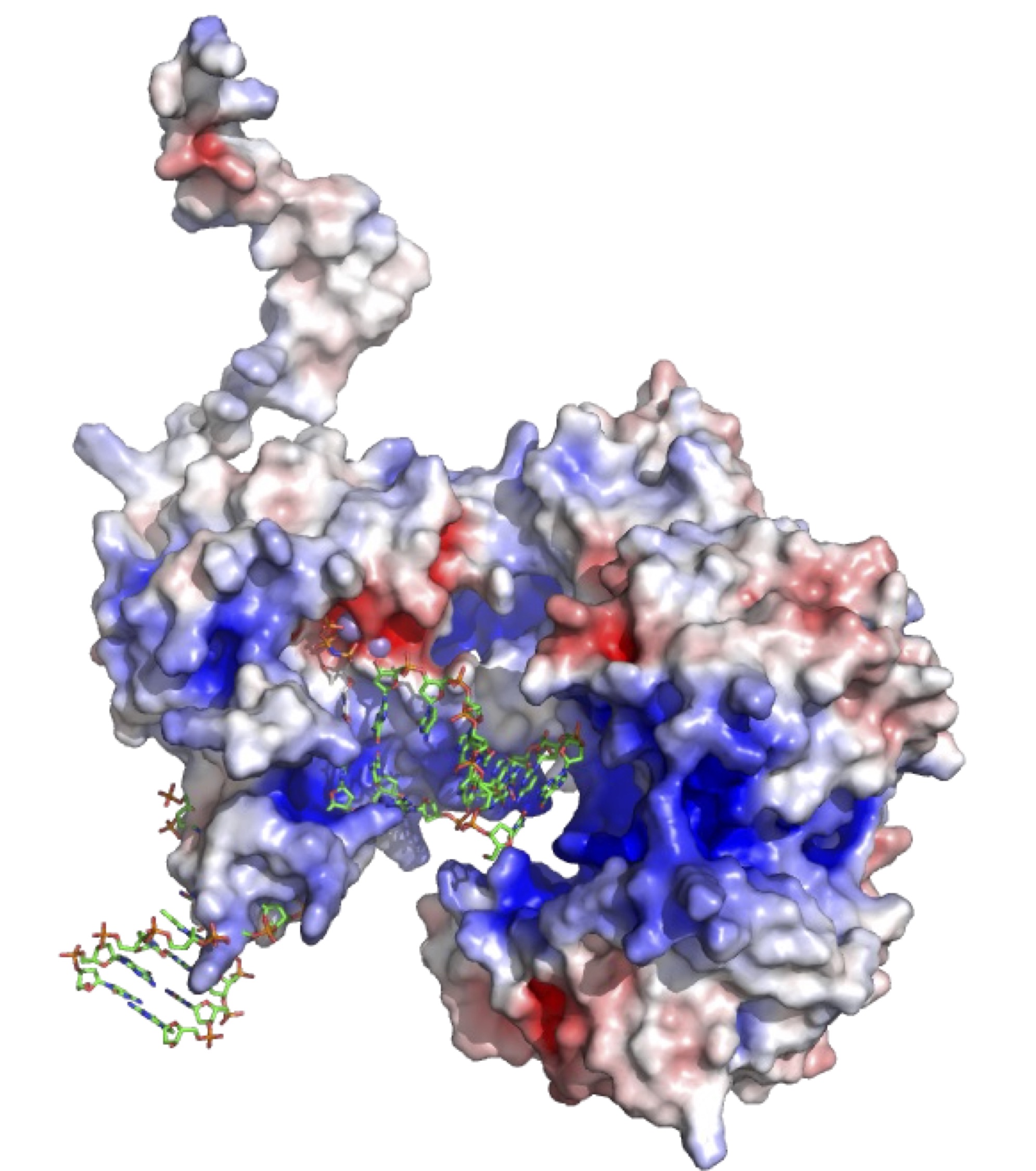
Recientemente hemos caracterizado una proteína denominada potenciador de escisión de secuencias de inserción, presente específicamente en la bacteria enteropatógena E. coli, y perteneciente a la superfamilia de las primasas arqueas/eucariotas. Nuestros estudios han demostrado que se trata de una nueva ADN polimerasa capaz de llevar a cabo la unión de extremos mediada por microhomología, que posee características bioquímicas típicas de las ADN polimerasas implicadas en la reparación de lesiones del ADN.
Miembros del grupo

Miguel de Vega José
Lab.: 405 Ext.: 4717
mdevega(at)cbm.csic.es

Alicia del Prado Díaz
Lab.: 405 Ext.: 4463
adelprado(at)cbm.csic.es

Silvia Díaz Arco
Lab.: 405 Ext.: 4463
silvia.diaz(at)cbm.csic.es

Lucía Alba Fernández
Lab.: 405 Ext.: 4463
lucia.alba(at)cbm.csic.es

Aitana García García
Lab.: 405 Ext.: 4463
Publicaciones representativas
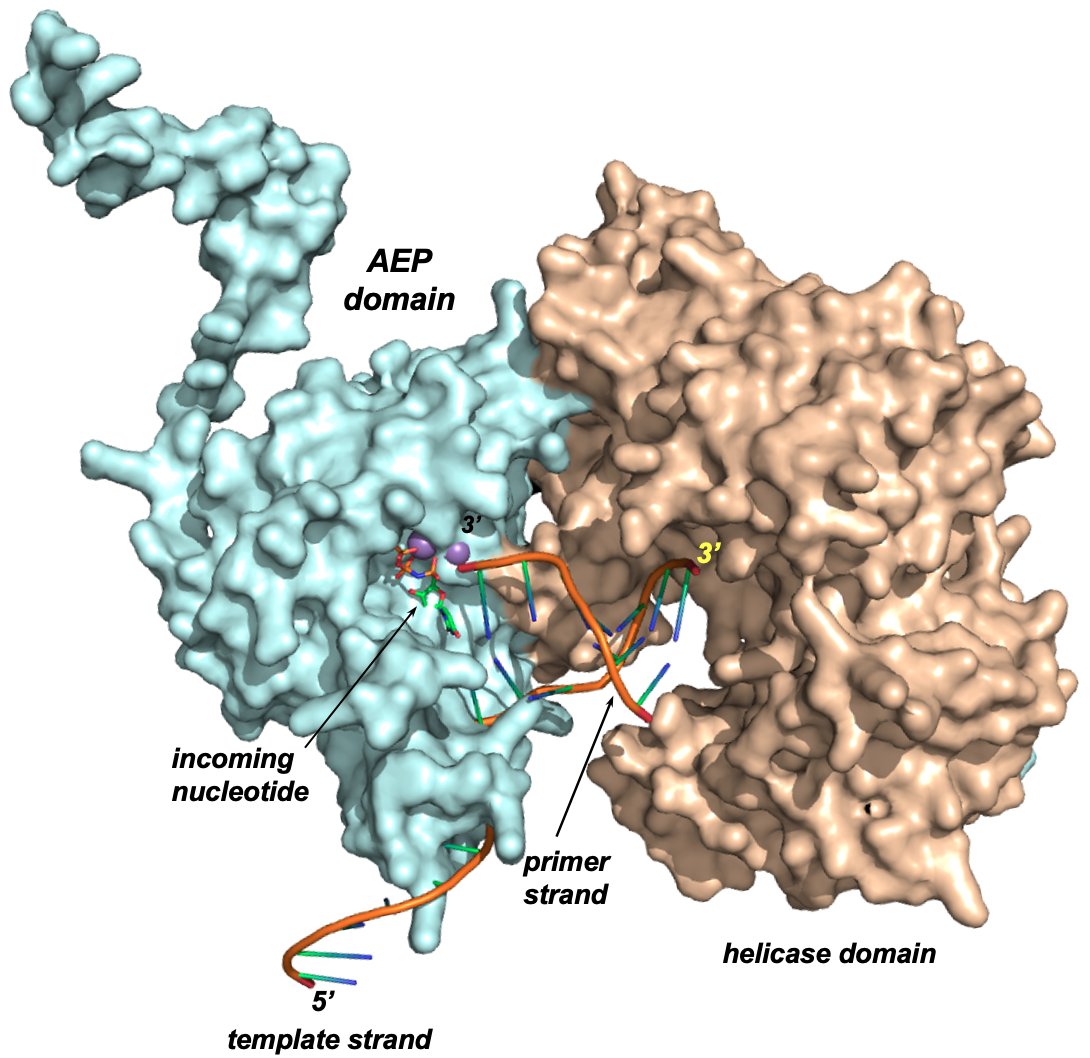
La proteína potenciadora de la inserción por escisión de secuencias de Escherichia coli enterohemorrágica
es una ADN polimerasa mediada por microhomología
ADN polimerasa con actividad de unión
mediada por microhomología
Patricia A. Calvo et al.
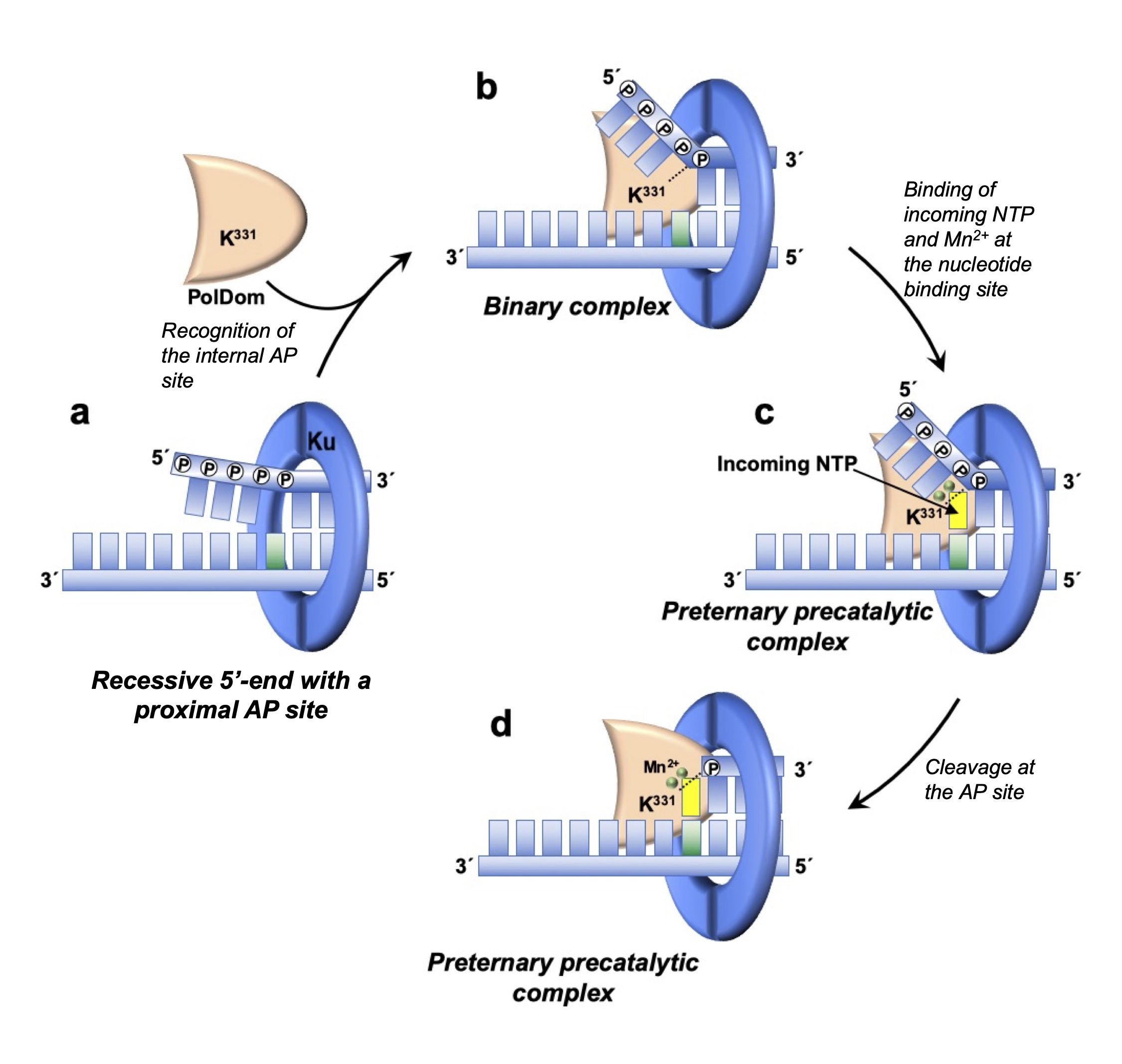
Bacterial Ligase D preternary-precatalytic complex performs efficient abasic sites processing at double strand breaks during nonhomologous end joining
Ana de Ory et al.
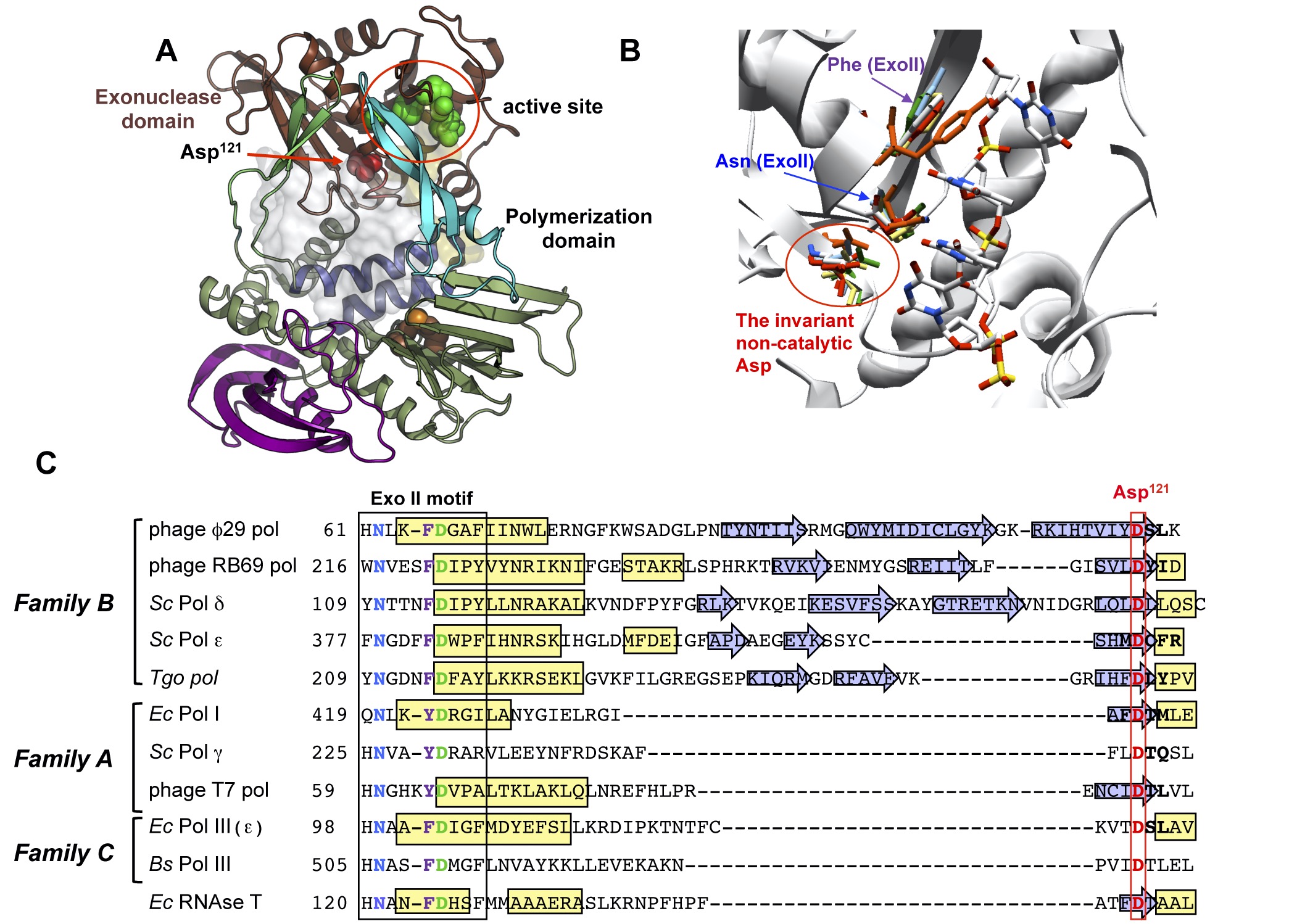
Noncatalytic aspartate at the exonuclease domain of proofreading DNA polymerases regulates both degradative and synthetic activities
Alicia del Prado et al.
Efficient processing of abasic sites by bacterial nonhomologous end-joining Ku proteins
Ana de Ory et al.