Programa Científico
Procesos fisiológicos y patológicos
UNIDADES EN ESTE PROGRAMA
Neuropatología molecular Redes metabólicas y señalizadoras en la enfermedad
GRUPO DE INVESTIGACIÓN
Estudios fisiopatológicos y desarrollo de terapias en modelos animales y celulares de enfermedades neurometabólicas
Realizamos investigación traslacional en enfermedades neurometabólicas raras. Generamos modelos animales y celulares personalizados para estudiar los mecanismos moleculares y fisiopatológicos subyacentes. Nuestro objetivo es desarrollar terapias personalizadas, incluyendo terapias específicas de mutación e intervenciones farmacológicas. Somos expertos en la generación de iPSCs y en el uso de CRISPR/Cas9 para modelar enfermedades in vitro e in vivo.
Investigación
El grupo pertenece al CIBER de Enfermedades Raras (CIBERER) y al Instituto de Investigación Sanitaria Hospital La Paz (IdiPAZ) y colabora activamente con el Centro de Diagnóstico de Enfermedades Moleculares (CEDEM, Facultad de Ciencias, UAM). Nuestra investigación se centra en las enfermedades neurometabólicas, acidemia propiónica (AP) e hiperfenilalaninemias (HPAs) entre otras, deficiencias enzimáticas de herencia autosómica recesiva, caracterizadas por la acumulación tóxica de precursores y la falta de metabolitos aguas abajo.
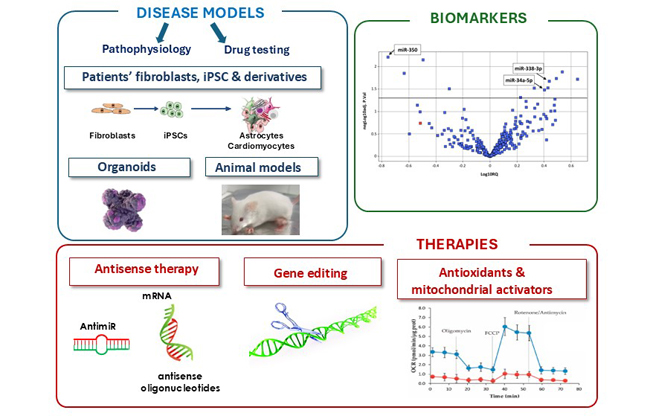
Nuestros proyectos representan la investigación traslacional con el objetivo de generar y caracterizar modelos animales y celulares relevantes para enfermedades específicas, que se utilizarán como herramientas de investigación para comprender los mecanismos moleculares y fisiopatológicos responsables de la enfermedad, analizar biomarcadores potenciales para el pronóstico y seguimiento e identificar nuevas dianas terapéuticas. El objetivo último es desarrollar terapias personalizadas, tanto enfoques específicos de mutación, como la edición génica y los oligonucleótidos antisentido, como terapias farmacológicas con compuestos antioxidantes y activadores mitocondriales, realizando estudios preclínicos en los modelos de enfermedad específicos.
El grupo de investigación tiene amplia experiencia en la generación de iPSCs a partir de fibroblastos de pacientes y su diferenciación a precursores neuronales, astrocitos y cardiomiocitos, linajes celulares relevantes para las enfermedades en estudio. También hemos utilizado ampliamente la tecnología de edición génica CRISPR/Cas9 para generar modelos celulares (células de hepatoma, iPSCs) y animales (ratón) de enfermedad, tanto knock-out como knock-in con mutaciones específicas del paciente.
Una línea de investigación implica el análisis de los mecanismos fisiopatológicos subyacentes a la APs, una de las acidemias orgánicas más frecuentes en la que hemos demostrado, utilizando un modelo de ratón, que la disfunción mitocondrial, el estrés oxidativo y la desregulación de miRNAs contribuyen a las complicaciones multiorgánicas de la enfermedad. Hemos revelado alteraciones en el mal manejo del Ca2+, asociadas a elevados niveles de ROS y mayor tasa de oxidación de SERCA2a, junto con la desregulación de cardiomiRs específicos, como mecanismos implicados en el desarrollo de cardiomiopatías asociadas a APs. Los estudios en cardiomiocitos derivados de iPSCs confirman estas alteraciones, y los cardiomiocitos de AP muestran una excitabilidad celular muy deprimida y un mayor riesgo de arritmias. En astrocitos de AP derivados de iPSCs observamos alteraciones de la función mitocondrial, desregulación de miRNAs y astrogliosis.
A lo largo de muchos años, el grupo ha estudiado la genética molecular de diferentes enfermedades metabólicas hereditarias, con un especial énfasis en variantes de tipo missense y de splicing. Hemos caracterizado el mecanismo molecular patogénico de muchas de estas variantes utilizando diversos sistemas de expresión procariotas y eucariotas y vectores reporteros de splicing denominados minigenes. Hemos desarrollado métodos para modular el proceso de splicing utilizando oligonucleótidos antisentido, habiendo corregido con éxito varios defectos de splicing, recuperando niveles normales de proteína y actividad, en modelos celulares de diferentes enfermedades. En la actualidad hemos implementado el uso de diferentes herramientas de edición génica (CRISPR/Cas, editores de bases) para corregir permanentemente variantes específicas causantes de enfermedades metabólicas hereditarias, tanto in vitro como in vivo.
Miembros del grupo

Lourdes Ruiz Desviat
Lab.: 220 Ext.: 4566/7830
lruiz(at)cbm.csic.es

Eva María Richard Rodríguez
Lab.: 220 Ext.: 4628/4596
erichard(at)cbm.csic.es

María del Mar Álvarez García
Lab.: 220 Ext.: 4596
malvarez(at)cbm.csic.es

Irene González Garnacho
Lab.: 220 Ext.: 4628
igonzalez(at)cbm.csic.es

Emilio Martínez García-Tenorio
Lab.: 220 Ext.: 4596
emilio.martinez(at)cbm.csic.es

Gonzalo Villena Navas
Lab.: 220 Ext.: 4596
gvillena(at)cbm.csic.es

José Vicente del Álamo Rodríguez
Lab.: 220 Ext.: 4596
jv.delalamo(at)cbm.csic.es

Laura Pons Renau
Lab.: 220 Ext.: 4596
lpons(at)cbm.csic.es
Publicaciones representativas
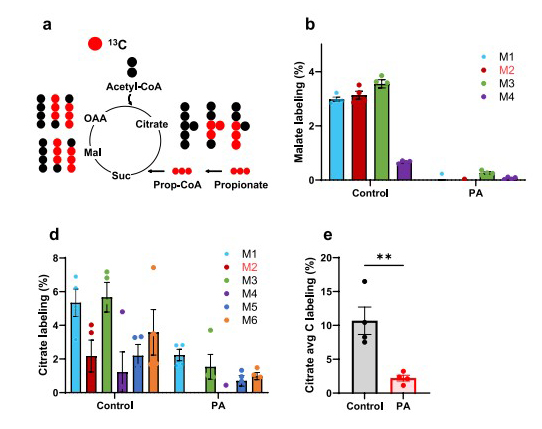
Metabolic flux analysis in hiPSC-CMs reveals insights into cardiac dysfunction in propionic acidemia
Eva Richard et al.
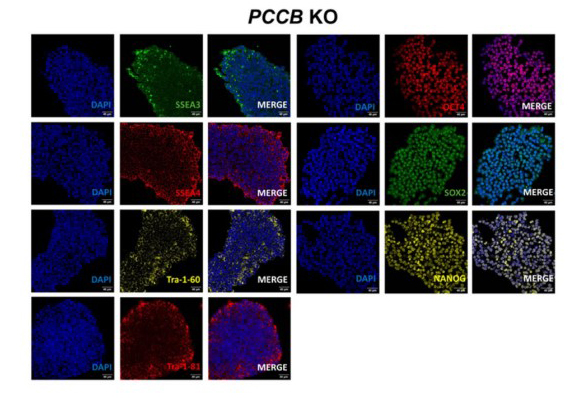
Novel CRISPR-Cas9 iPSC knockouts for PCCA and PCCB genes: advancing propionic acidemia research
Emilio M. García-Tenorio et al.
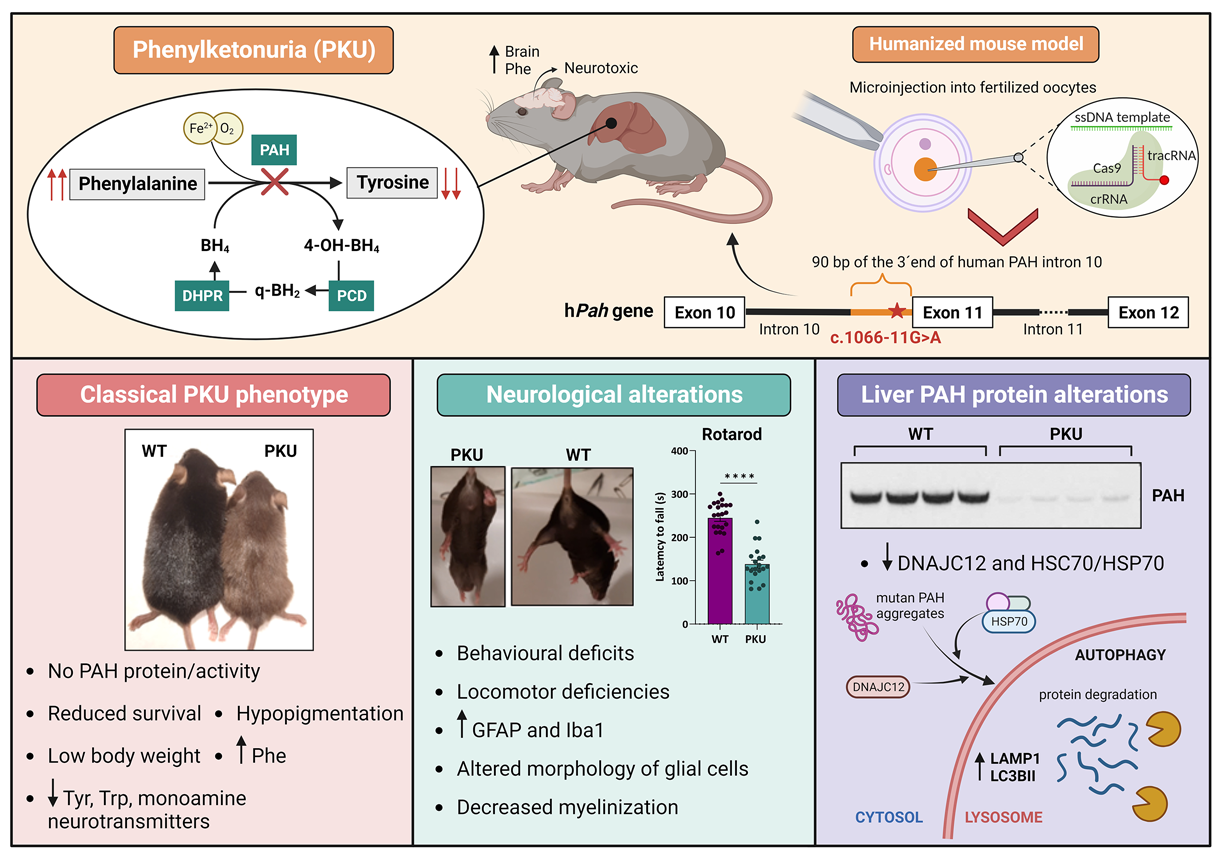
PAH deficient pathology in humanized c.1066-11G>A phenylketonuria mice
Ainhoa Martínez-Pizarro et al.
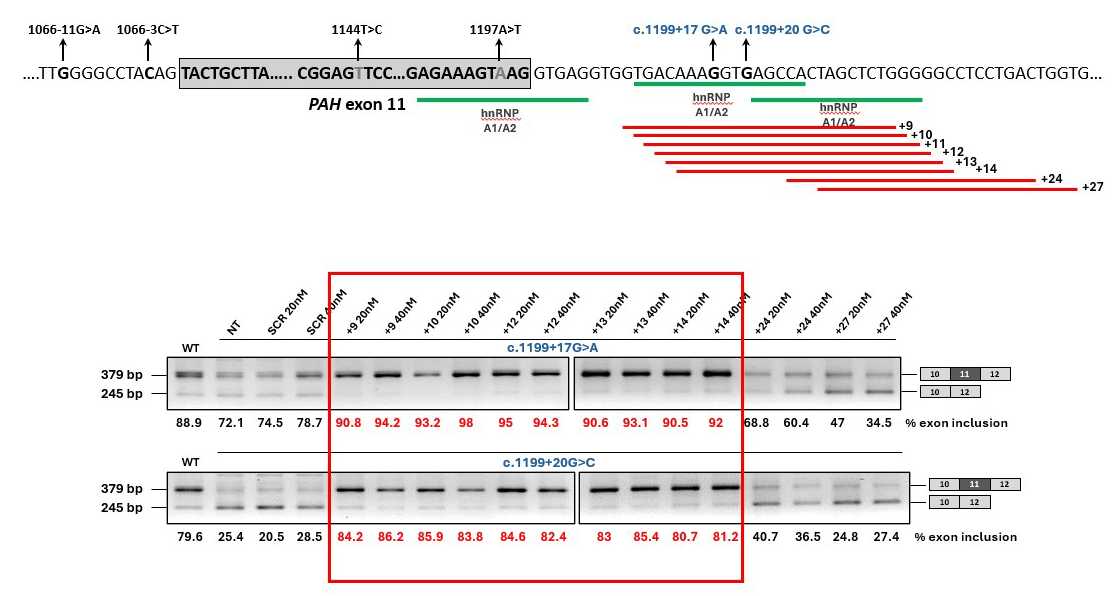
Splice-Switching Antisense Oligonucleotides Correct Phenylalanine Hydroxylase Exon 11 Skipping Defects and Rescue Enzyme Activity in Phenylketonuria
Ainhoa Martínez-Pizarro et al.