Programa Científico
Dinámica y función del genoma
GRUPO DE INVESTIGACIÓN
Regulación de la traducción en eucariotas y su implicación en la fisiopatología de células y organismos

Iván Ventoso Bande
Regulación de la traducción en eucariotas y su papel en la homeostasis celular.
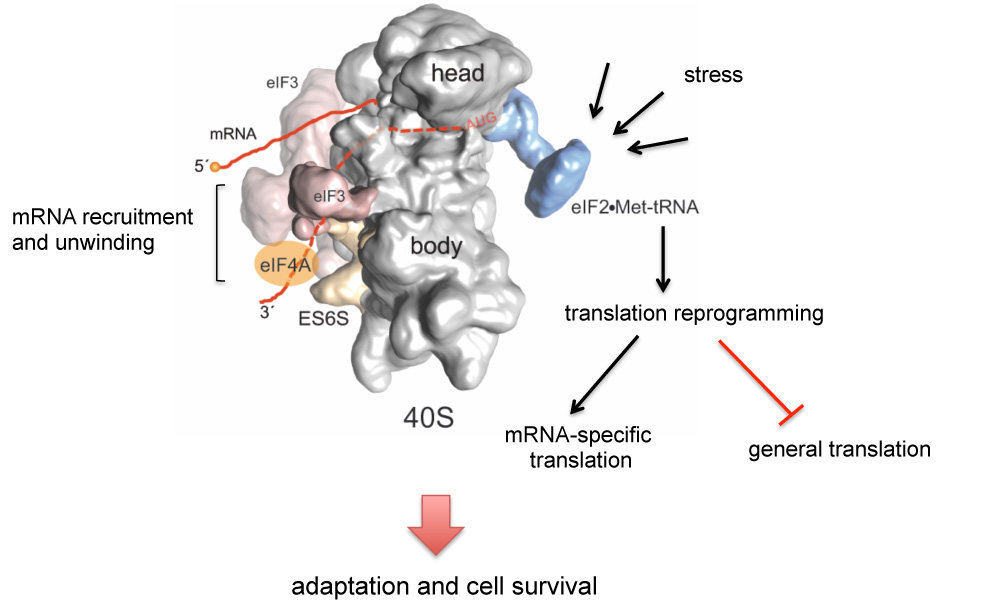
Investigamos cómo los sistemas eucarióticos regulan la traducción, tanto a nivel global como de manera específica de mRNA. Nuestro objetivo es identificar nuevos elementos reguladores presentes en los mRNAs y en los ribosomas que jueguen un papel relevante en el control traduccional durante la proliferación celular, la respuesta al estrés y la supervivencia en modelos de células humanas y levaduras. Estudiamos cómo la reprogramación traduccional a través de la fosforilación del factor de iniciación de la traducción eIF2 en respuesta al estrés, modula la proteostasis y la autofagia celular. Para ello, combinamos el análisis genético, el modelado estructural, la cuantificación de la expresión génica a tiempo real y el análisis masivo de traducción mediante perfiles de polisomas y secuenciación de ARN. También estudiamos cómo algunos virus interfieren con la traducción celular mediante proteínas virales que bloquean parcialmente la función del ribosoma, lo que constituye una poderosa herramienta de trabajo.
Investigación
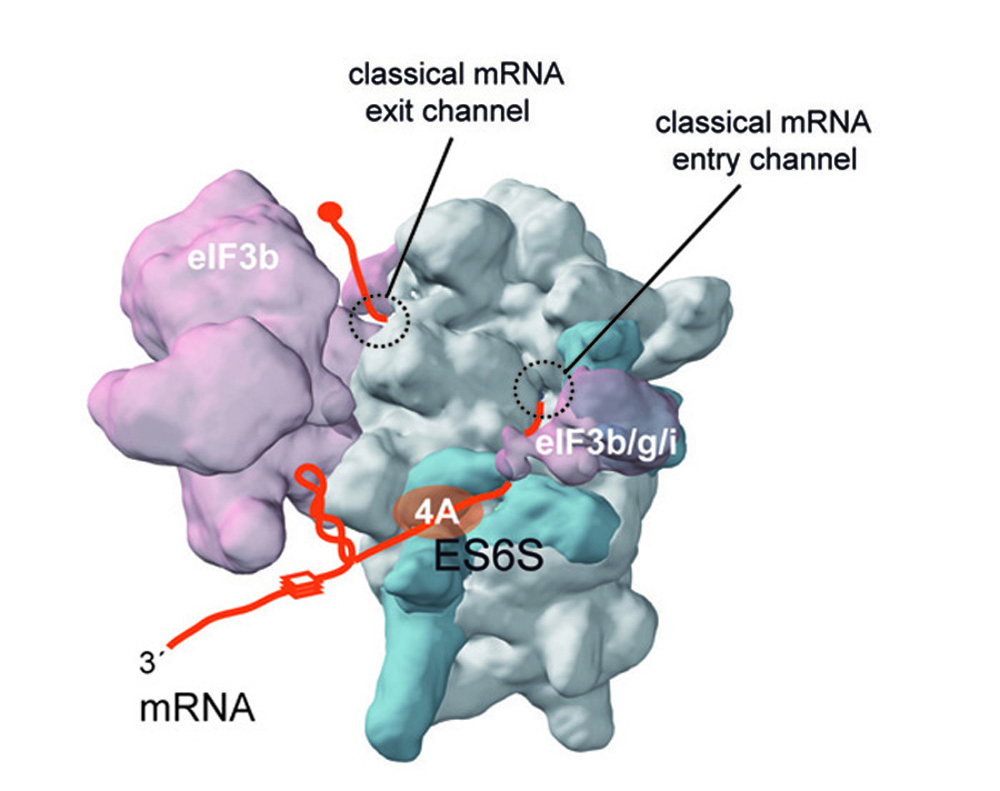
Hemos identificado la región ES6S de la subunidad ribosómica 40S como la puerta de entrada del ARNm y el escaneo del PIC 43S durante el inicio de la traducción. Nuestros datos sugieren que la región ES6S podría estar sirviendo como plataforma para el reclutamiento de helicasas de ARN (eIF4A y DDX3, entre otras) implicadas en el desenrollamiento de la estructura secundaria del ARN. El bloqueo de la región ES6S afectó de manera diferente a la traducción de los ARNm que codifican algunos protooncogenes (H-Ras, CCND3, ODC-1, etc.), genes implicados en la transducción de señales con ARNm 5´UTR largos y estructurados, y también algunos ARNm virales. Actualmente estamos evaluando la región ES6S como diana para moléculas antitumorales y antivirales.
Más recientemente, hemos descubierto que el efecto diferencial de la proteína no estructural 1 (NSP1) del SARS-CoV-2 sobre la traducción depende de la composición del 5′ UTR y del sesgo en el uso de codones del ARNm diana. Nuestros datos sugieren la existencia de dos mecanismos para el reclutamiento del ARNm a la subunidad 40S (enhebrado y encaje) y una interacción funcional entre la velocidad de elongación (sesgo de codones) y el inicio de la traducción. También estamos estudiando las implicaciones de nuestros resultados para la evolución molecular del SARS-CoV-2 y otros virus respiratorios humanos.
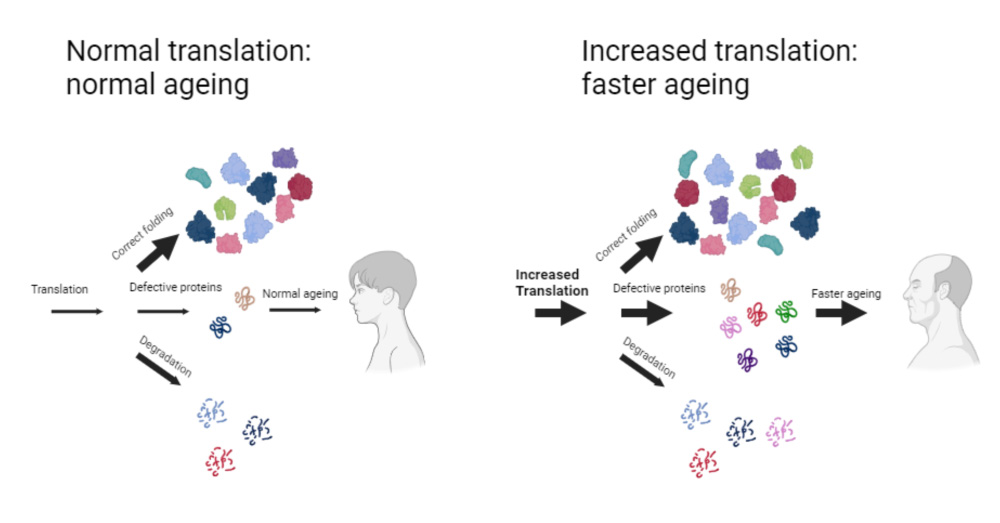
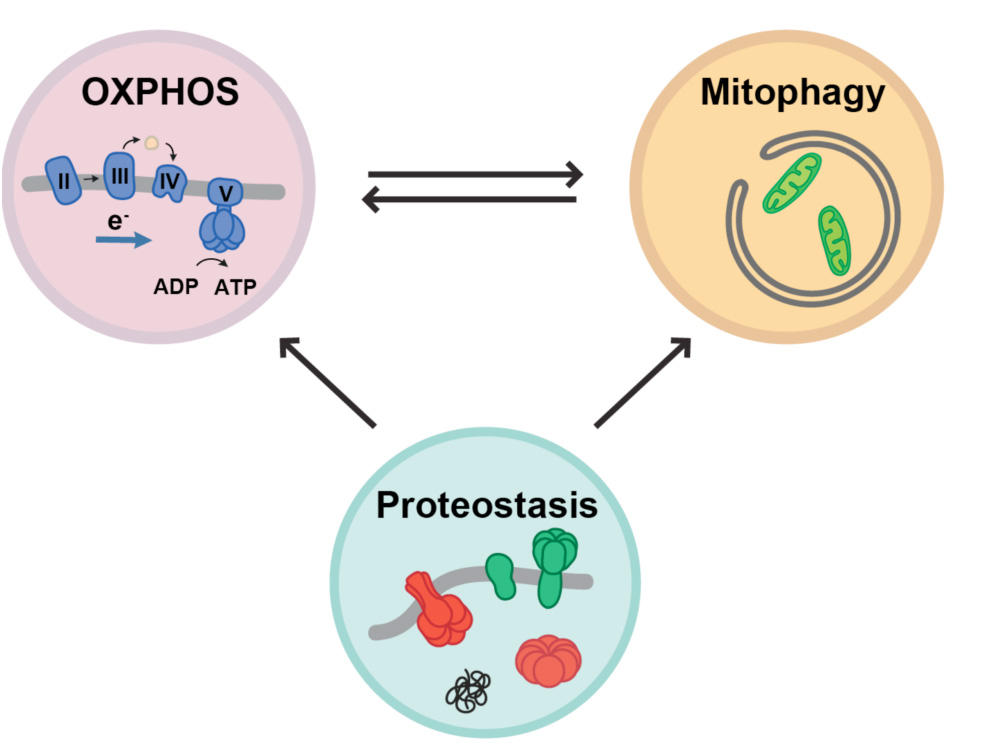
Continuamos estudiando las relaciones moleculares y funcionales entre respuesta al estrés, reprogramación traduccional y homeostasis celular. Hemos encontrado que las levaduras incapaces de fosforilar eIF2α en respuesta al estrés, no sólo pierden su capacidad de responder a éste, sino que también exhiben un envejecimiento acelerado debido a una disfunción de la proteostasis y autofagia. Hemos identificado algunos genes relacionados con la actividad del proteasoma cuya expresión se regula a través de la fosforilación de eIF2, y estamos analizando su papel en procesos que regulan la proteostasis celular como la agregación de proteínas, autofagia, mitofagia y su efecto sobre supervivencia.
Miembros del grupo

Juan José Berlanga Chiquero
Lab.: 106 Ext.: 4714/4534
jberlanga(at)cbm.csic.es

Iván Ventoso Bande
Lab.: 106/114.4( despacho) Ext.: 4809
iventoso(at)cbm.csic.es

Miguel Angel Rodríguez Gabriel
Lab.: 106 Ext.: 4816
marodriguez(at)cbm.csic.es

Margarita Cabrera Sola
Lab.: 106 Ext.: 4534
mcabrera(at)cbm.csic.es

Mercedes Núñez Bayón
Lab.: 106 Ext.: 4534
mnunnez(at)cbm.csic.es

Carla Henares Sánchez
Lab.: 106 Ext.: 4809
carla.henares(at)cbm.csic.es

Amaia Zamora Reyes
Lab.: 106 Ext.: 4809
Publicaciones representativas
An mRNA-binding channel in the ES6S region of the translation 48S-PIC promotes RNA unwinding and scanning
Irene Díaz-López et al.
Antagonistic effects of mitochondrial matrix and intermembrane space proteases on yeast aging
Montserrat Vega et al.
Translational control of gene expression by eIF2 modulates proteostasis and extends lifespan
Tamara Jiménez-Saucedo et al.
Título
Autores