Programa Científico
Interacciones con el entorno
UNIDADES EN ESTE PROGRAMA
Microorganismos en la salud y el bienestar Desarrollo y función del sistema inmunitario
GRUPO DE INVESTIGACIÓN
Modelado molecular

Paulino Gómez-Puertas
Laboratorio de biología computacional. El trabajo se centra en la integración de información evolutiva y estructural para estudiar la función de las proteínas, la simulación de procesos dinámicos de interacción proteína-proteína y proteína-ligando, el desarrollo de nuevos sistemas de diseño de fármacos «in silico» y la generación de nuevos métodos cuantitativos para biología computacional.
Investigación
Proyectos en curso:
– Análisis mediante simulación computacional de reacciones enzimáticas catalizadas por enzimas de interés biomédico. Diseño de inhibidores específicos. Cohesina: Análisis de las interacciones moleculares entre los componentes proteicos del anillo de cohesina y de la interacción del complejo proteico con el ADN. Carbapenemasas: Simulación dinámica de la interacción de carbapenemasas bacterianas (VIM-2, KPC-2, OXA-48) con sustratos e inhibidores conocidos.
– Desarrollo de un nuevo y eficaz sistema de diseño de fármacos basado en la simulación dinámica computacional de estructuras macromoleculares. Basado en el análisis de centros activos de enzimas, el método desarrollado por nuestro grupo consiste en simular estas estructuras proteicas mediante dinámica molecular durante varios cientos de nanosegundos, seleccionar diferentes estructuras representativas y filtrar una base de datos de compuestos 3D para cada una de ellas. Se utiliza actualmente en el diseño de nuevos inhibidores de carbapenemasas y el diseño de inhibidores específicos como fármacos antitumorales.
– Desarrollo de métodos eficaces para calcular trayectorias de valores de parámetros mínimos o máximos (p.e., trayectorias de mínima energía) a través de superficies de cualquier número de dimensiones.
Miembros del grupo

Paulino Gómez Puertas
Lab.: 313 Ext.: 4663
pagomez(at)cbm.csic.es

Iñigo Marcos Alcalde
Lab.: 313 Ext.: 4662
imarcos(at)cbm.csic.es
Publicaciones representativas
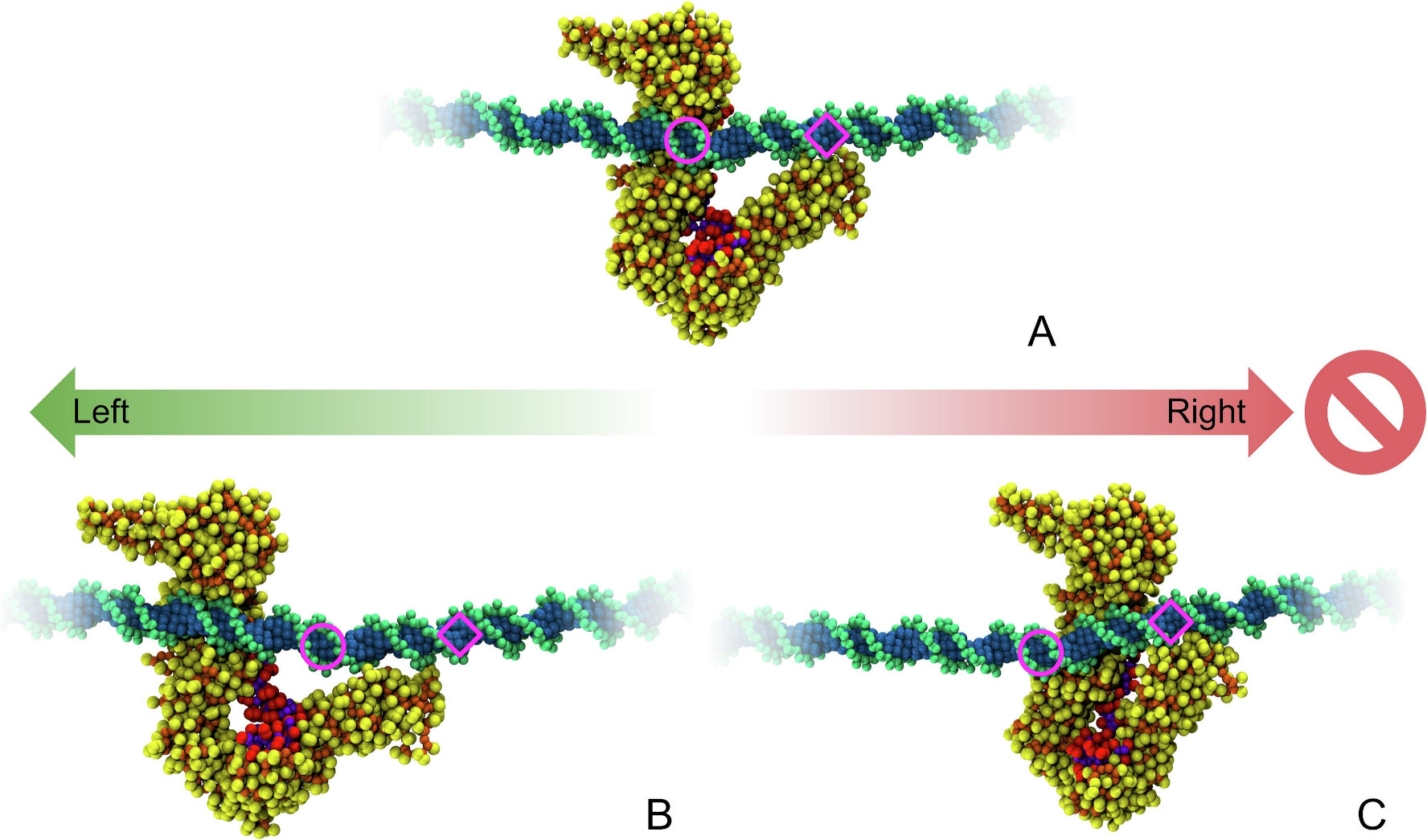
STAG2-RAD21 Complex: a Unidirectional DNA Ratchet Mechanism in Loop Extrusion
David Ros-Pardo et al.
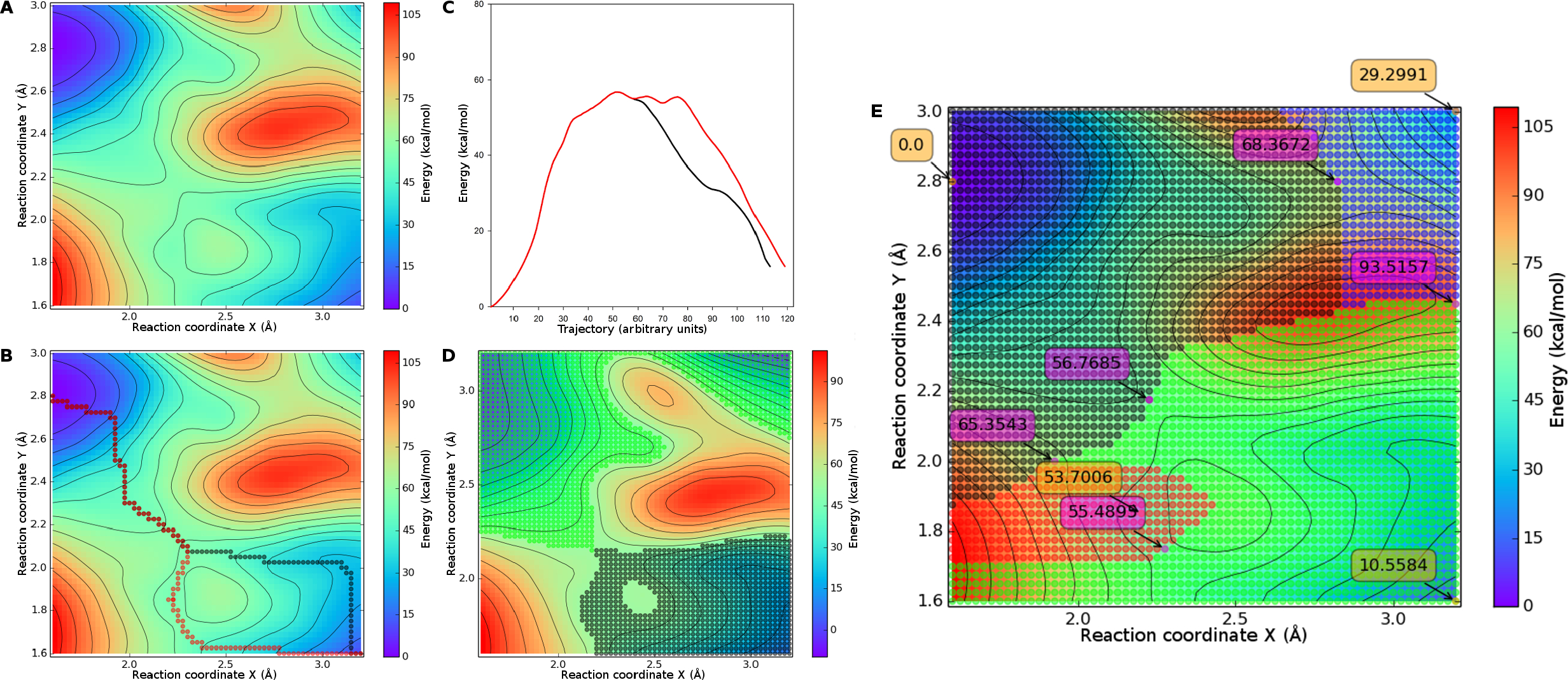
MEPSA: minimum energy pathway analysis for energy landscapes
Iñigo Marcos-Alcalde et al.
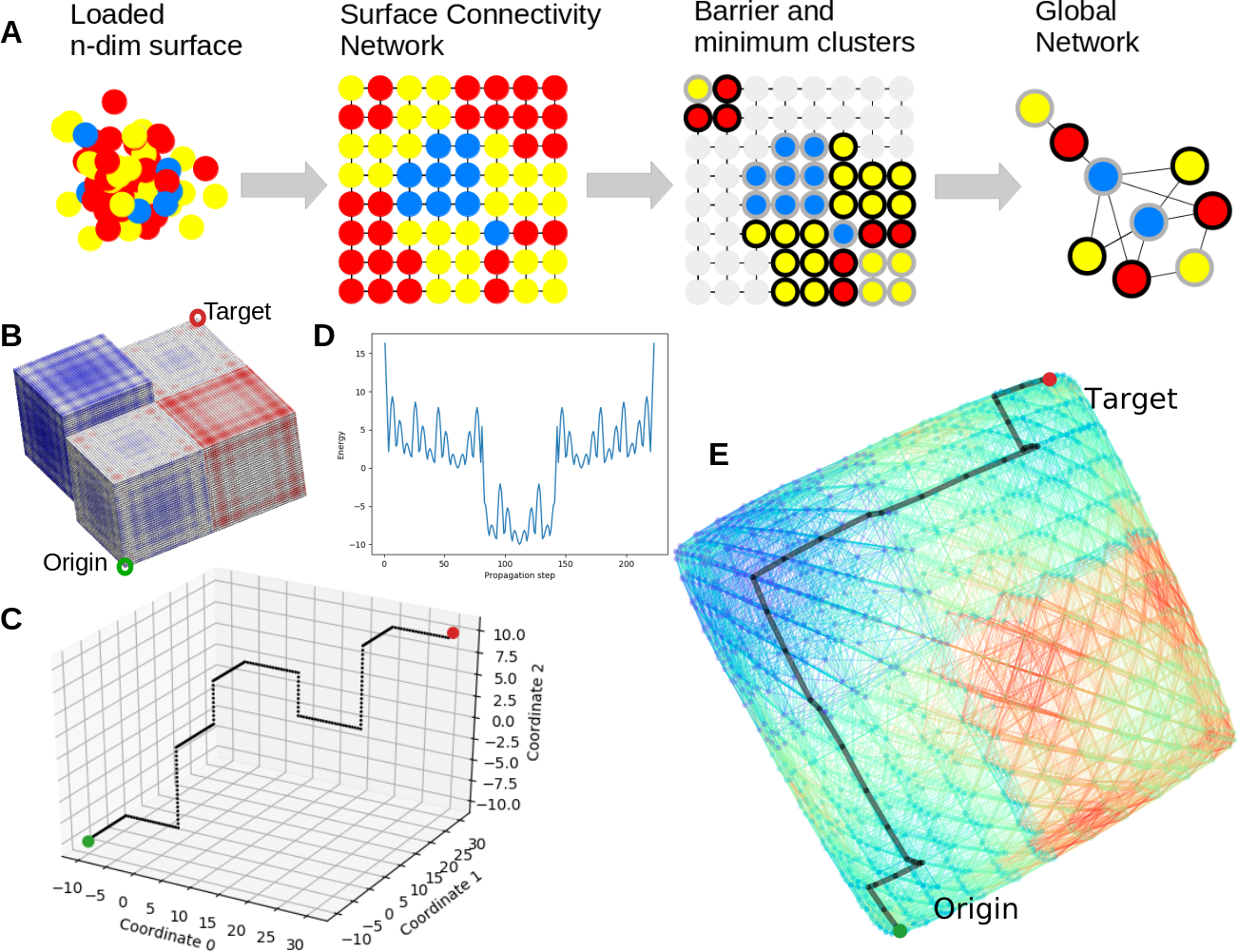
MEPSAnd: minimum energy path surface analysis over n-dimensional surfaces
Iñigo Marcos-Alcalde et al.
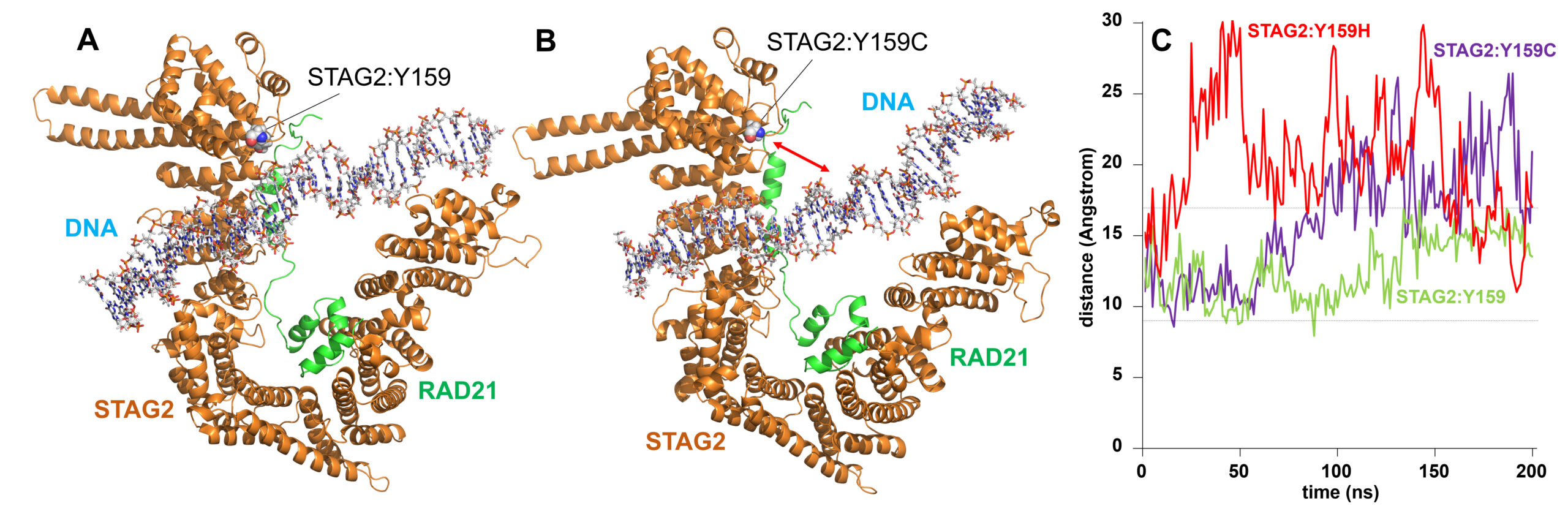
STAG2: Computational Analysis of Missense Variants Involved in Disease
David Ros-Pardo et al.