Programa Científico
Interacciones con el entorno
UNIDADES EN ESTE PROGRAMA
Microorganismos en la salud y el bienestar Desarrollo y función del sistema inmunitario
GRUPO DE INVESTIGACIÓN
Modulación de la inmunidad antiviral por proteasas virales y RNAs no codificantes

Margarita Sáiz Zalabardo
Nuestro grupo está interesado en descifrar los mecanismos moleculares desarrollados por virus RNA para inactivar la respuesta antiviral del hospedador, en especial los que implican la actividad de proteasas virales. La interacción de los genomas y proteínas virales con sensores celulares es esencial para la activación de una respuesta inmune eficaz y puede determinar el desarrollo de enfermedad. Trabajamos también en la caracterización de regiones de RNA viral no-codificantes y su aplicación como antivirales basados en RNA.
Investigación
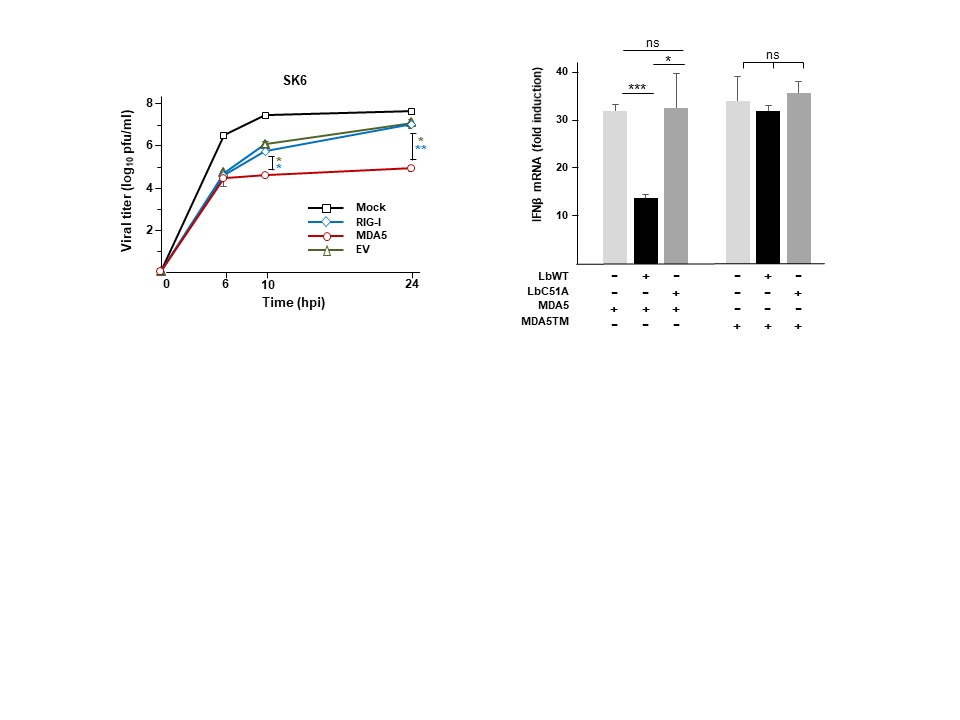
Nuestro interés se centra en el estudio de las interacciones entre virus RNA y las células hospedadoras con implicación de las regiones no-codificantes (NCRs) de los genomas virales y la actividad catalítica de las proteasas virales. Nuestros estudios incluyen la identificación de proteínas sensoras de la infección y presencia de genomas virales de RNA así como la caracterización de los mecanismos moleculares de evasión inmune utilizados por los virus para combatir la respuesta antiviral del hospedador basada en la inducción de interferón (IFN) tipo I en las células infectadas. El resultado del equilibrio entre la respuesta antiviral y el antagonismo viral puede determinar el desarrollo de enfermedad y la patogénesis. Usamos el virus de la fiebre aftosa (FMDV), un patógeno altamente infeccioso de distribución mundial, como modelo y estamos trabajando activamente en la identificación de proteínas efectoras de inmunidad innata como posibles dianas de las proteasas virales codificadas por el genoma de FMDV (Lpro y 3Cpro) y su posible interferencia con diferentes rutas de señalización relevantes en la respuesta a la infección por virus RNA. Hemos descrito un nuevo mecanismo de evasión viral basado en la proteólisis de LGP2 y MDA5, sensores inmunes de la familia de los RIG-I-like receptors, por la proteasa Leader (Lpro) de FMDV y la degradación redundante de cGAS por 3Cpro y Lpro. Estas interacciones impactan negativamente en la inmunidad antiviral, favoreciendo la infección.
El grupo también trabaja en la caracterización de los dominios estructurales presentes en las 5´ y 3´NCRs del genoma de FMDV, incluyendo interacciones RNA-RNA, RNA-proteínas y requerimientos estructurales implicados en viabilidad viral, capacidad infectiva y estimulación de respuesta inmune innata. Nuestros estudios previos sobre las NCRs de FMDV nos llevaron a explorar las aplicaciones bioterapéuticas de RNAs no-codificantes sintéticos derivados de las 5´ y 3´NCRs de FMDV (ncRNAs) demostrando su capacidad de inducir una actividad antiviral de amplio espectro y de potenciar la respuesta inmune específica mediada por células B y T inducida por vacunación. Hemos descrito el efecto inhibidor de los ncRNAs frente a la infección por patógenos virales de distintas familias incluyendo virus zoonóticos relevantes (FMDV, West Nile virus, Rift Valley fever virus, African swine fever virus) y mostrado su actividad biológica en células animales de especies ganaderas y salvajes, y también in vivo en ratón y cerdo. La actividad anti-pan-coronavirus de los ncRNAs ha sido probada recientemente mostrando la actividad profiláctica intrínseca del RNA desnudo frente a SARS-CoV-2 en un modelo de COVID-19 en ratón. Estamos trabajando también en el desarrollo de nuevas formulaciones basadas en nanotecnología para mejorar la vehiculización de los ncRNAs. Los resultados derivados de nuestros estudios contribuirán a incrementar el conocimiento sobre la interacción entre patógenos virales y el sistema de inmunidad innata de la célula hospedadora y al diseño de estrategias nuevas y más eficaces frente a virus circulantes y otros que puedan emerger en el futuro. Aprender cómo los virus neutralizan la respuesta inmune del hospedador identificando específicamente proteínas efectoras relevantes como dianas puede contribuir también al diseño de nuevas estrategias terapéuticas frente a interferonopatías de tipo I.
Miembros del grupo

Margarita Sáiz Zalabardo
Lab.: 105 Ext.: 4729
msaiz(at)cbm.csic.es

Miguel Ramón Rodríguez Pulido
Lab.: 105 Ext.: 4522
mrrodriguez(at)cbm.csic.es

Catalina Grabowski Pinto
Lab.: 105 Ext.: 4522
cgrabowski(at)cbm.csic.es

Miryam Polo Hernández
Lab.: 105 Ext.: 4522
mpolo(at)cbm.csic.es
Publicaciones representativas
Non-coding RNAs derived from the foot-and-mouth disease virus genome trigger broad antiviral activity against coronaviruses
Miguel Rodriguez Pulido et al.
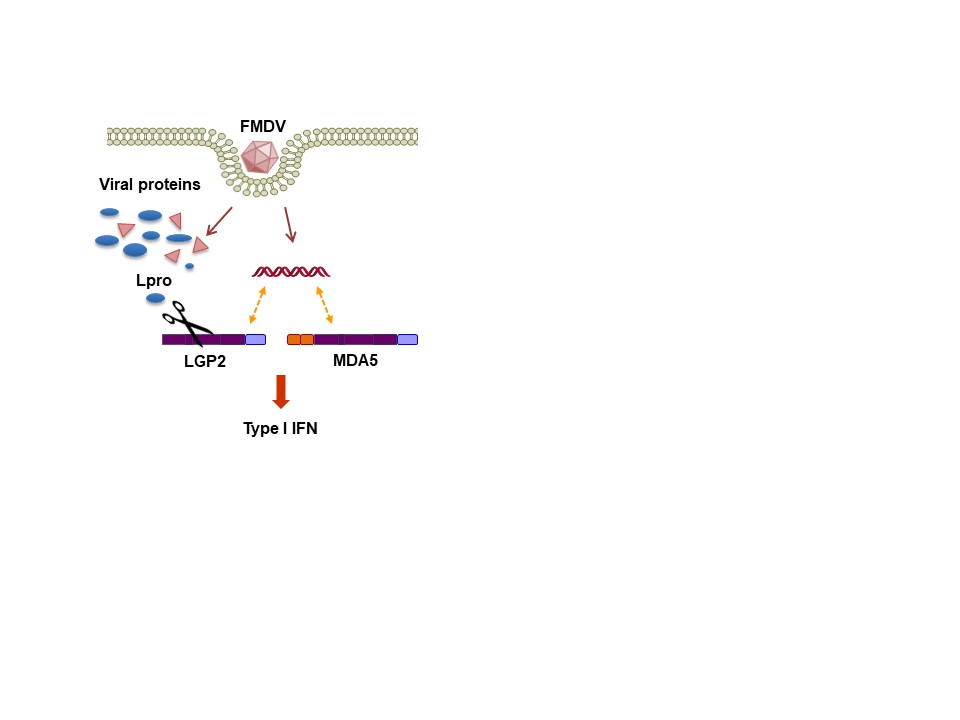
Innate immune sensor LGP2 is cleaved by the Leader protease of foot-and-mouth disease virus
Miguel Rodríguez Pulido et al.
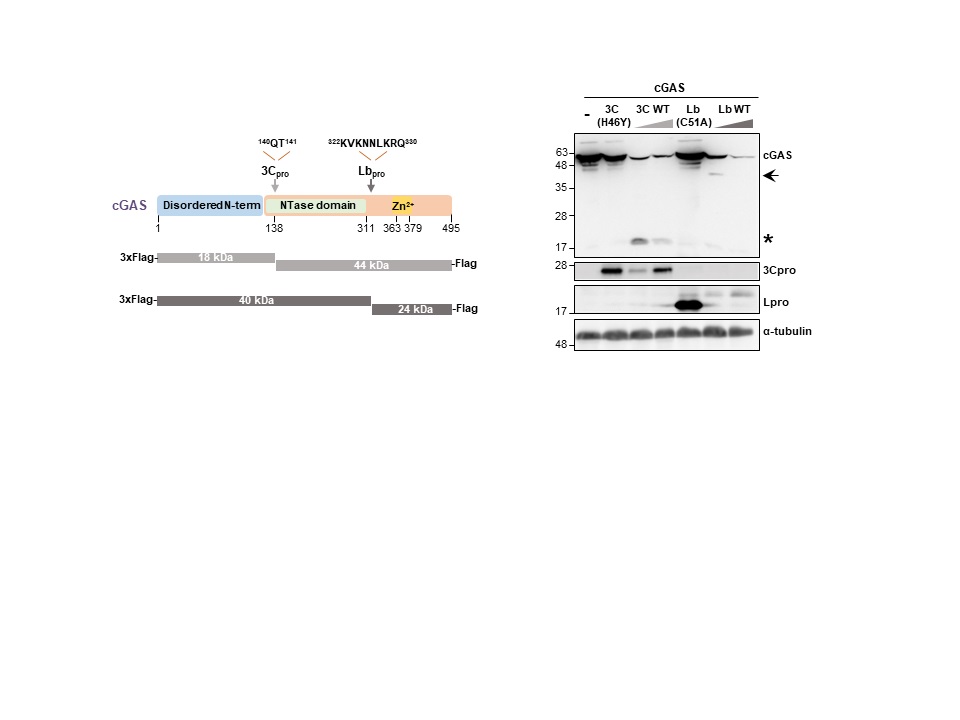
The antiviral response triggered by the cGAS/STING pathway is subverted by the foot-and-mouth disease virus proteases
Miguel Ángel Sanz et al.

MDA5 cleavage by the Leader protease of foot-and-mouth disease virus reveals its pleiotropic effect against the host antiviral response
Miguel Rodríguez Pulido et al.