Regulación de la traducción en eucariotas y su implicación en la fisiopatología de células y organismos
Resumen de Investigación:
Investigamos cómo los sistemas eucarióticos regulan la traducción, tanto a nivel global como de manera específica de mRNA, para tratar de identificar nuevos elementos reguladores (estructuras y secuencias) presentes en los ribosomas y en los mRNA, y nuevas actividades de los factores de iniciación de la traducción (eIFs) que puedan ser relevantes en la traducción diferencial de mRNAs durante la proliferación celular y la respuesta al estrés. Hemos descubierto que la región ES6S de la subunidad ribosómica 40S actúa como puerta de entrada del mRNA durante el proceso de scanning del complejo de preiniciación, promoviendo la eliminación de la estructura secundaria del mRNA por helicasas de RNA como el factor eIF4A que se asocian a la región ES6S. El bloqueo de la región ES6S afectó de manera diferencial a la traducción de mRNAs con 5'UTR largas y complejas, como las presentes en algunos mRNAs que codifican protooncogenes (H-Ras, CCND3, ODC-1, etc.) y otros genes implicados en señalización celular. En estos momentos estamos evaluando la actividad anti-tumoral de algunos oligonucleótidos y aptámeros dirigidos contra la región ES6S de la subunidad ribosómica 40S.
Queremos entender mejor la reprogramación traduccional que se observa en respuesta al estrés en modelos eucarióticos (levaduras y ratón) y que es dependiente de la actividad de los factores eIF2 y eIF2A, así como su impacto fisiológico a nivel de organismo, incluyendo el estudio de parámetros de supervivencia y envejecimiento en estos modelos. Recientemente hemos encontrado que las levaduras incapaces de fosforilar eIF2α en respuesta al estrés, no sólo pierden su capacidad de responder a éste, sino que también exhiben un envejecimiento acelerado debido a una disrupción de la proteostasis celular.
También estudiamos cómo los Alfavirus han evolucionado para adaptar la traducción de sus mRNAs a las condiciones de estrés presentes en las células y tejidos infectados, un modelo que nos está ayudando a entender mejor la respuesta antiviral en mamíferos y que nos proporciona un marco conceptual para desarrollar virus oncoselectivos para su uso en viroterapia de tumores humanos.
Usamos varios organismos modelo, y combinamos técnicas de genética molecular, análisis estructural de RNA y ribosomas, modelos animales y técnicas de análisis masivo, junto con la Bioinformática y la Biología de sistemas.

Figura 1. Nube de palabras con los temas que investigamos en el laboratorio
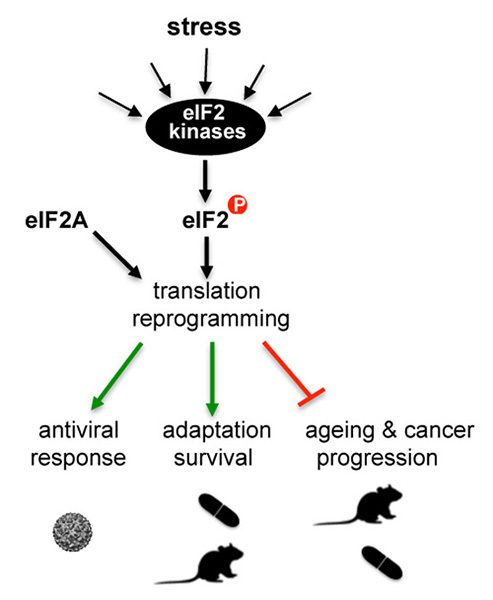
Figura 2. La respuesta al estrés iniciada por la fosforilación de eIF2α promueve una atenuación general de la síntesis de proteínas, y simultáneamente, un incremento de la traducción de un grupo de mRNAs (≈ 300 en humanos) implicados en la respuesta al estrés. Ambos eventos son necesarios para coordinar una respuesta adaptativa al estrés, que también puede modular procesos más a largo plazo como la supervivencia y el envejecimiento del organismo. Algunos de estos mRNAs se traducen de una manera no canónica y requieren otros factores de traducción alternativos como el eIF2A.
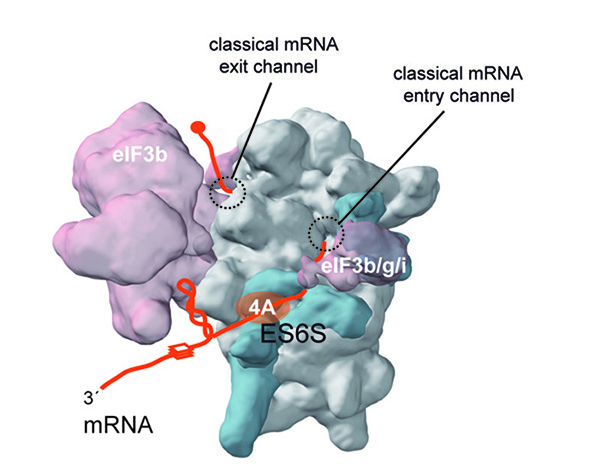
Figura 3. Modelo del complejo de preiniciación de la traducción (48S-PIC) durante el proceso de scanning, mostrando cómo se enhebra el mRNA en la región ES6S de la subunidad 40S para ser desenrollado por la helicasa eIF4A. Se señalan algunos elementos de estructura en el 5'UTR del mRNA como horquillas y G-quadruplex (cajas). Por simplicidad, no se muestra el resto de componentes del complejo eIF4F.

| Apellidos | Nombre | Laboratorio | Ext.* | Categoría profesional | |
|---|---|---|---|---|---|
| Alcalde García | José | 106 | 4534 | jalcalde(at)cbm.csic.es | E.Ayudantes De Invest. De Los Oo.Publicos De Investigacion |
| Berlanga Chiquero | Juan José | 106 | 4714/4534 | jberlanga(at)cbm.csic.es | Profesor Titular Universidad, GA |
| Jiménez Saucedo | Tamara | 106 | 4534 | tjimenez(at)cbm.csic.com | Titulado Sup.de Actividades Técn. y Profes. GP1 |
| Rodríguez Gabriel | Miguel Ángel | 106 | 4816 | marodriguez(at)cbm.csic.es | Profesor Titular Universidad, GA |
| Ventoso Bande | Iván | 126/114.4( despacho) | 4809 | iventoso(at)cbm.csic.es | Profesor Titular Universidad, GA |
Publicaciones relevantes:
- Díaz-López I, Toribio R, Berlanga JJ, Ventoso I (2019) An mRNA-binding channel in the ES6S region of the translation 48S-PIC promotes RNA unwinding and scanning. Elife. 8. doi: 10.7554/eLife.48246.
- Toribio R, Díaz-López I, Boskovic J, Ventoso I (2018) Translation initiation of alphavirus mRNA reveals new insights into the topology of the 48S initiation complex. Nucleic Acids Res. 46(8): 4176-4187. doi: 10.1093/nar/gky071.
- Martín R, Berlanga JJ, de Haro C (2013) New roles of the fission yeast eIF2α kinases Hri1 and Gcn2 in response to nutritional stress. J Cell Sci. 126: 3010-3020.
- Matia-Gonzalez AM, Hasan A, Moe, GH, Mata J, Rodriguez-Gabriel MA (2013) Functional characterization of Upf1 targets in Schizosaccharomyces pombe. RNA Biol. 10: 1057-1065.
- Toribio R, Ventoso I (2010) Inhibition of host translation by virus infection in vivo. Proc Natl Acad Sci U S A. 107(21): 9837-42. doi:10.1073/pnas.1 004110107.
- Ventoso I, Sanz MA, Molina S, Berlanga JJ, Carrasco L, Esteban M (2006) Translational resistance of late alphavirus mRNA to eIF2alpha phosphorylation: a strategy to overcome the antiviral effect of protein kinase PKR. Genes Dev. 20(1):87-100.
- Berlanga JJ, Ventoso I, Harding HP, Deng J, Ron D, Sonenberg N, Carrasco L, de Haro C (2006) Antiviral effect of the mammalian translation initiation factor 2alpha kinase GCN2 against RNA viruses. EMBO J. 25(8):1730-40.
Tesis doctorales:
- Tamara Jiménez Saucedo. Role of eIF2α-dependent translational regulation in aging. Dpto. Biología Molecular. UAM 2020. Sobresaliente Cum Laude.
- Irene Díaz López. Involvement of ES6S region of 40S subunit in mRNA threading and scanning during translation initiation. Dpto. Biología Molecular. UAM 2019. Sobresaliente Cum Laude.
- Marina Portantier. Papel del complejo Ccr4-Not en la respuesta a estrés mediada por la MAPK Spc1 en Schizosaccharomyces pombe. Dpto. Biología Molecular. UAM 2013. Sobresaliente Cum Laude.
- Javier del Pino García. Interacción funcional de la eIF2a quinasa GCN2 con el virus de la inmunodeficiencia humana VIH-1. Dpto. Biología Molecular. UAM 2012. Sobresaliente Cum Laude.
- Ruth Martín Martín. Caracterización funcional de las eIF2α quinasas de Schizosaccharomyces pombe en distintas situaciones de estrés. Dpto. Biología Molecular. UAM 2012. Sobresaliente Cum Laude.
- Ana María Matía González. Role of Upf1 and Csx1 on posttranscriptional regulation of gene expression in Schizosaccharomyces pombe. Dpto. Biología Molecular. UAM 2011. Sobresaliente Cum Laude.
- René Toribio López. Cambios Traduccionales que regulan la interacción virus hospedador. Implicación en el desarrollo de virus oncolíticos. Dpto. Biología Molecular. UAM 2010. Sobresaliente Cum Laude.
- Jael Sotelo Álvarez. Wip1: una nueva proteína moduladora de la diferenciación sexual en Schizosaccharomyces pombe. Dpto. Microbiología II. UCM 2010. Sobresaliente Cum Laude.
- Isabela Alonso González. Caracterización funcional de la elF2α quinasa GCN2 de células de mamífero. Dpto. Biología Molecular. UAM 2009. Sobresaliente Cum Laude.




















