Replicación del DNA del bateriófago ø29
Resumen de Investigación:
Durante más de 50 años, desde su fundación en 1967, nuestro grupo ha estudiado los mecanismos moleculares de la replicación del bacteriofago Φ29, desde una perspectiva multidisciplinar. Bajo el liderazgo científico y personal de la profesora Margarita Salas, se han desentrañado multiples aspectos bioquímicos y genéticos del ciclo de vida del virus y se han desarrollado aplicaciones biotecnológicas a partir de los resultados obtenidos.
Sin ninguna duda, la pérdida de la Dra. Salas es irreemplazable. Sin embargo, estamos decididos a realizar el esfuerzo necesario, no solo para mantener su legado sino también para completar los proyectos de investigación actuales. La excelencia y el rigor científico que Margarita nos inculcó nos ayudarán para finalizar esta tarea, que consideramos es la mejor manera de la que sus discípulos podemos rendirle homenaje.
Obituario de Margarita Salas en Nature
Principales líneas de investigación actuales.
- Nuevos aspectos de la replicación del DNA del bacteriófago Φ29 in vitro e in vivo.
Esta línea de investigación incluye un estudio detallado de la localización subcelular de la replicación viral en Bacillus subtilis, su hospedador natural, así como nuevos avances en la caracterización molecular de la maquinaria replicativa del virus, mediante experimentos in vitro. - Los betatectivirus como nuevo modelo de replicación de genomas lineales iniciados por proteína terminal.
Los Tectivirus son virus bacterianos icosaédricos, sin cola y que contienen una membrana interna bajo la cápsida y pueden infectar bacterias Gram-negativas (Alfatectivirus) o Gram-positivas (Beta- y Gammatectivirus). Este grupo de virus ha cobrado relevancia en los últimos años, gracias a diversos estudios sobre su relevancia ecológica y evolutiva. Hemos caracterizado recientemente la DNA polimerasa viral y el mecanismo de iniciación de la replicación mediada por proteína terminal del virus Bam35, virus modelo del género Betatectivirus. Actualmente, nuestro trabajo se enfoca en la caracterización de proteínas virales de unión al DNA, tales como una SSB (proteína de unión a DNA de cadena sencilla), que podría realizar un papel esencial en la replicación del genoma viral.
Adicionalmente, en colaboración con Annika Gillis y Jacques Mahillon (Université Catholique de Louvain, Bélgica), estamos caracterizando el mecanismo de replicación de un nuevo Betatectivirus. - Nuevas actividades y aplicaciones biotecnológicas de DNA polimerasas virales.
En los últimos 10 años, en colaboración con Miguel de Vega (CBMSO, CSIC), nuestro laboratorio ha desarrollado variantes de la DNA polimerasa de Φ29 con mayor velocidad y capacidad de amplificación y con mejor termorresistencia. Como continuación de este trabajo previo, otro de los objetivos en los que trabajamos actualmente es en la obtención de variantes de esta DNA polimerasa que permitan la amplificación de DNAs en diferentes condiciones.
La caracterización de la DNA polimerasa de Bam35 (B35DNAP), nos permitió encontrar un nuevo enzima con posibles aplicaciones biotecnológicas. Al igual que la DNA polimerasa de Φ29, la B35DNAP replica el DNA de forma fiel, procesiva y acoplada al desplazamiento de la hebra no replicada. Además, esta nueva DNA polimerasa es capaz de replicar moldes de ADN dañados (capacidad TLS, del inglés translesion synthesis). Actualmente estamos trabajando en la obtención de variantes que permitan amplificar muestras en mal estado de conservación. - Estudio global del interactoma proteico virus-huésped mediante el empleo de doble híbrido acoplado a análisis mediante secuenciación masiva.
El desarrollo reciente de métodos de alta capacidad permite el estudio de los sistemas biológicos desde un punto de vista global, en el que todos y cada uno de sus elementos son analizados. De esta forma, se pueden analizar por ejemplo todas las interacciones proteína-proteína (PPI) en un determinado modelo biológico mediante la modificación de métodos ya conocidos como el doble híbrido de levaduras (Y2H). Mediante la aplicación de esta metodología estamos analizando de manera global las PPI por primera vez en un modelo virus-huésped formado por un bacteriófago-bacteria, en concreto, sistema formado por el tectivirus atemperado Bam35 y su hospedador natural Bacillus thuringiensis HER1410. Esta metodología nos permitirá caracterizar en más detalle la estirpe y las relaciones virus-hospedador. - Encapsulación de la maquinaria de transcripción.
En colaboración con Paco Monroy (Universidad Complutense de Madrid) y dentro del consorcio: Nanobiocargo, estamos trabajando en la encapsulación en vesículas lipídicas de la maquinaria molecular necesaria para la transcripción del DNA de Φ29. Estudiaremos la energía que se necesita para este proceso, así como el calor que se disipa. Para ello utilizaremos técnicas de microscopía y pinzas ópticas.
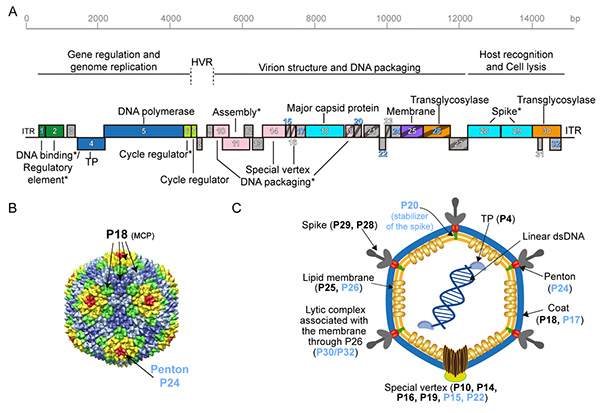
Figura 1. El genoma del virus Bam35 y estructura de la partícula viral (Modificado de Berjón-Otero et al., 2017). A. Mapa genético del fago Bam35. Los rectángulos corresponden a los ORF predichos en el genoma Bam35.. B. Modelo de la capside del virus Bam35, basado en la estructura cristalográfica del Alfatectivirus PRD1. C. Modelo propuesto para la partícula viral Bam35. Representación esquemática de la partícula viral Bam35 en la que se indica la localización propuesta o conocida de cada una de sus proteínas.

| Apellidos | Nombre | Laboratorio | Ext.* | Categoría profesional | |
|---|---|---|---|---|---|
| Lechuga Mateo | Ana | 409 | 4463 | a.lechuga(at)cbm.csic.es | Titulado Sup. Actividades Tecn. y Prof.GP1 |
| Muruzabal Galarza | Ane | 409 | 4463 | Estudiante TFM | |
| Ordoñez Cencerrado | Carlos David | 409 | 4463 | cordonez(at)cbm.csic.es | Titulado Sup. Actividades Tecn. y Prof.GP1 |
| Prado Díaz | Alicia del | 409 | 4463 | adelprado(at)cbm.csic.es | Titulado Sup. Actividades Tecn. y Prof.GP1 |
| Redrejo Rodríguez | Modesto | 409 | 4463 | mredrejo(at)cbm.csic.es | Estancia/Investigador |
Publicaciones relevantes:
- de Vega, M., et al., Improvement of Φ29 DNA Polymerase Amplification Performance by Fusion of DNA Binding Motifs. PNAS, 2010. 107(38): p. 16506-11. DOI: 10.1073/pnas.1011428107
- Berjón-Otero, M., et al., DNA Polymerase from Temperate Phage Bam35 Is Endowed with Processive Polymerization and Abasic Sites Translesion Synthesis Capacity. PNAS, 2015. 112(27): p. E3476-84. DOI: 10.1073/pnas.1510280112
- Mojardin, L. and M. Salas, Global Transcriptional Analysis of Virus-Host Interactions between Phage Phi29 and Bacillus Subtilis. J Virol, 2016. 90(20): p. 9293-304. DOI: 10.1128/JVI.01245-16
- Berjón-Otero, M., et al., Disclosing Early Steps of Protein-Primed Genome Replication of the Gram-Positive Tectivirus Bam35. NAR, 2016. 44(20): p. 9733-9744. DOI: 10.1093/nar/gkw673
- Berjón-Otero, M., et al., Bam35 Tectivirus Intraviral Interaction Map Unveils New Function and Localization of Phage Orfan Proteins. J Virol, 2017. 91(19). DOI: 10.1128/JVI.00870-17
- Redrejo-Rodríguez, M., et al., Primer-Independent DNA Synthesis by a Family B DNA Polymerase from Self-Replicating Mobile Genetic Elements. Cell Rep, 2017. 21(6): p. 1574-1587. DOI: 10.1016/j.celrep.2017.10.039
- van Nies, P., et al., Self-Replication of DNA by Its Encoded Proteins in Liposome-Based Synthetic Cells. Nat Commun, 2018. 9(1): p. 1583. DOI: 10.1038/s41467-018-03926-1
- del Prado, A., et al., Noncatalytic Aspartate at the Exonuclease Domain of Proofreading DNA Polymerases Regulates Both Degradative and Synthetic Activities. PNAS, 2018. 115(13): p. E2921-E2929. DOI: 10.1073/pnas.1718787115
- del Prado, Rodríguez I, Lázaro JM, Moreno-Morcillo M de Vega M, Salas M (2019). New insights into the coordination between the polymerisation and 3´-5´exonuclease activities in f29 DNA polymerase. Scientific Reports. 2019 Jan 29;9(1):923. DOI: 10.1038/s41598-018-37513-7
- de la Torre, I., et al., Tyrosines Involved in the Activity of Phi29 Single-Stranded DNA Binding Protein. PLoS One, 2019. 14(5): p. e0217248. DOI: 10.1371/journal.pone.0217248
- del Prado, A., et al., The Loop of the TPR1 Subdomain of Phi29 DNA Polymerase Plays a Pivotal Role in Primer-Terminus Stabilization at the Polymerization Active Site. Biomolecules, 2019. 9(11). DOI: 10.3390/biom9110648
Tesis doctorales:
- Mónica Berjón Otero (UAM, 2017) Caracterización molecular del bacteriófago BAM35. Mecanismo de replicación del DNA y estudio del interactoma proteico. Directores: Margarita Salas y Modesto Redrejo Rodríguez.
- Eugenia Santos Del Río (UAM, 2017) Papel del motivo LExE de las DNA polimerasas que inician con proteína terminal en la interacción con el nucleótido entrante. Estabilización de los sustratos en el centro activo de polimerización mediada por el subdominio TPR1. Directores: Margarita Salas y Miguel de Vega.
Patentes:
- Variantes de la ADN Polimerasa del Bacteriófago Phi29 con termoactividad mejorada. Solicitud de Patente ES201531910A (2015), CSIC y CRG.
- Primer-Independent DNA Polymerases and Their Use for DNA Synthesis, Solicitud de pantente ES201731236A (2017), CSIC e Instituto Pasteur




















