Metabolismo en cáncer y envejecimiento
El linfoma difuso de células B grandes (DLBCL) es la neoplasia linfoide maligna más común en adultos y representa un grupo heterogéneo de tumores con distintos subtipos que difieren en alteraciones genéticas, resultado clínico, respuesta al tratamiento y pronóstico. Aproximadamente del 5 al 15% de los DLBCL son linfomas de células B de alto grado (HGBL) con reordenamientos de MYC y BCL2 y se conocen como linfoma "double hit" (DHL) o linfoma “triple hit” (THL) si incluyen reordenamientos en BCL6. Debido a la activación simultánea de estos oncogenes, los DHL se encuentran entre el subtipo de linfoma más agresivo y quimiorresistente, con opciones de tratamiento mínimas. Igualmente, la co-expresión de las proteínas MYC y BCL2 sin reordenamientos subyacentes se considera un nuevo indicador de pronóstico adverso denominado linfoma de doble expresión (DEL). La expresión desregulada de MYC contribuye a la reprogramación metabólica de las células tumorales a través de una compleja red de factores. Específicamente, las células B del linfoma transformado con c-MYC dependen del metabolismo de la glutamina para sus procesos bioenergéticos. Además, el metabolismo de la serina, a través de la actividad de la enzima SHMT2, contribuye a la biología de los linfomas agresivos con desregulación de MYC. Sin embargo, las características metabólicas de los linfomas HGBL-DH no se conocen en detalle y podrían implicar el descubrimiento de potenciales vulnerabilidades. Comprender la biología única de estos tumores, incluido su metabolismo, podría ser de utilidad para explorar terapias dirigidas más allá del enfoque actual en los inhibidores de BCL2 y MYC.
Nuestro laboratorio, que abrió sus puertas en septiembre de 2021, se centra en caracterizar las propiedades metabólicas de estos tumores en particular, utilizando análisis de expresión transcripcional y metabolómica en modelos preclínicos y muestras de pacientes, y detectar nuevas vulnerabilidades metabólicas que podrían usarse como nuevas terapias dirigidas para estos linfomas de alto grado con muy mala respuesta a las terapias estándar.
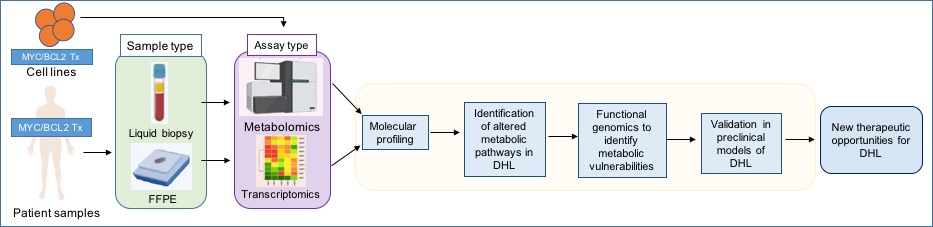
Figura 1. Esquema de la investigación desarrollada en el laboratorio

|
Apellidos |
Nombre |
Laboratorio |
Extensión |
|
Categoría Profesional |
|
Ortega Molina |
Ana |
408 |
4509 |
Investigador |
|
|
Jimeno Lumeras |
Rebeca |
408 |
4574 |
M3 | |
|
Prieto Carro |
Cristina |
408 |
4574 |
M2 | |
| López Martín-Lunas | Beatriz | 408 | 4574 | blopezml@cbm.csic.es | M2 |
| Salazar Ortego | Sara | 408 | 4574 | sara.salazaro@estudiante.uam.es | Beca JAE intro |
| González Pérez | Marta | 408 | 4574 | martgo78@ucm.es | Estudiante fin de máster |
1.- Ortega-Molina, A and Efeyan, A. (2021). From mouse genetics to targeting the Rag GTPase pathway. Molecular & Cellular Oncology 8 (5): 1979370. doi: 10.1080/23723556.2021.1979370
2.- Ortega-Molina, A*, Lebrero-Fernández, C., Sanz, A., Deleyto-Seldas, N, Plata-Gómez A.B., Menéndez, C., Graña-Castro O., Caleiras, E., Efeyan, A*. (2021). Inhibition of Rag GTPase signaling in mice suppresses B cell responses and lymphomagenesis with minimal detrimental tradeoffs. Cell Reports, 36(2):109372. doi: 10.1016/j.celrep.2021.109372
3.- De la Calle Arregui C., Plata-Goméz A.B., Deleyto-Seldas N., García F., Ortega-Molina A., Abril-Garrido J., Rodriguez E., Nemazanyy I., Tribouillard L., de Martino A., Caleiras E., Campos-Olivas C., Mulero M., Laplante M., Muñoz J., Pende M., Sabio G., Sabatini D.M., Efeyan A. (2021). Limited survival and impaired hepatic fasting metabolism in mice with constitutive Rag GTPase signaling. Nature Communications, 12(1):3660. doi: 10.1038/s41467-021-23857-8
4.- Parsa, S#., Ortega-Molina, A#, Ying HY., Jiang, M., Teater, M., Wang, J., Zhao, C., Reznik, E., Pasion, JP., Kuo, D., Mohan, P., Wang, S., Camarillo, JM., Thomas, PM., Jain, N., Garcia-Bermudez, J., Cho, B., Tam, W., Kelleher, NL., Socci, N., Dogan, A., De Stanchina, E., Ciriello, G., Green, M., Li, S., Birsory, K., Melnick, AM., Wendel, HG. (2020). The serine hydroxymethyltransferase-2 (SHMT2) initiates lymphoma development through epigenetic tumor suppressor silencing. Nature Cancer, 1: 653-664. doi: 10.1038/s43018-020-0080-0
5.- Ortega-Molina A., Deleyto-Seldas N., Carreras J. Sanz, A., Lebrero-Fernandez, C., Menendez, C., Vandenberg, A., Fernandez-Ruiz, B., Marin-Arraiza, L., de la Calle Arregui, C., Plata-Gomez, AB., Caleiras, E., de Martino, A., Martinez-Martin, N., Troule, K., Piñeiro-Yañez, E., Nakamura, Y., Araf, S., Victora, GD., Okosun, J., Fitzgibbon, J., Efeyan, A. (2019). Oncogenic Rag GTPase ignaling enhances B cell activation and drives follicular lymphoma sensitive to pharmacological inhibition of mTOR. Nature Metabolism, 1 (8): 775-789. doi: 10.1038/s42255-019-0098-8
6.- Jiang Y#, Ortega-Molina A#, Geng H#, Ying HY#, Hatzi K, Parsa S, McNally D, Wang L, Doane AS, Agirre Ena X, Teater M, Meydan C, Li Z, Poloway D, Wang S, Ennishi D, Scott DW, Stengel KR, Kranz JE, Holson E, Sharma S, Young JW, Chu CS, Roeder RG, Shaknovich R, Hiebert SW, Gascoyne RD, Tam W, Elemento O, Wendel HG, Melnick AM. (2017). CREBBP Inactivation Promotes the Development of HDAC3 Dependent Lymphomas. Cancer Discovery, 7(1): 38-53. doi: 10.1158/2159-8290.CD-16-0975
7.- Ortega-Molina A.#, Boss I. W.#, Canela A.#., Pan H., Jiang Y., Zhao C., Jiang M., Hu D., Agirre X., Niesvixky I., Lee J.-E., Chen H.-T., Ennishi D., Scott D., Mottok A., Hother C., Liu S., Cao X.-J., Tam W., Shaknovich R., Garcia B. A., Gascoyne R., Ge K., Shilatifard A., Elemento O., Nussenzweig A., Melnick A. M., Wendel H.G. (2015). The histone lysisne methyltransferase KMT2D sustains a gene expression program that represses B cell lymphoma development. Nature Medicine, 21(10): 1199-1208. doi: 10.1038/nm.3943
*Co-corresponding authors
# Co-first author




















