Descubrimiento y mejora de proteínas para aplicaciones biotecnológicas mediante métodos de (ultra)alta capacidad
Resumen de Investigación:
La diversidad microbiana es un vasto reservorio de información genética valorizable mediante su aplicación industrial, desde clusters biosintéticos de moléculas bioactivas hasta genes que codifiquen enzimas para biocatálisis. La sinergia entre la microbiología y la nanotecnología para desarrollar nuevos métodos de cribado es clave para encontrar enzimas de manera más rápida y eficiente, permitiendo que los laboratorios académicos desarrollemos campañas de cribado hasta ahora limitadas a grandes empresas.
En nuestro laboratorio utilizamos selecciones biológicas o métodos de cribado de ultra-alta capacidad basados en microfluídica para encontrar genes y enzimas de interés en la diversidad natural o artificial. Las selecciones biológicas acoplan la mejora en una propiedad de interés a la supervivencia de un huésped bajo presión de selección. Utilizando este y otros métodos de ingeniería de proteínas hemos desarrollado variantes estables y solubles de enzimas para biocatálisis, como esterasas, deshalogenasas, además de proteínas fluorescentes para aplicaciones de localización in vivo a altas temperaturas.
Sin embargo, la complejidad del metabolismo celular limita la aplicación de las selecciones biológicas. La microfluídica permite la miniaturización de los ensayos, con una reducción de 1000x en coste y volumen, manteniendo velocidades de cribado de varios miles de individuos por segundo. Por lo tanto, el uso de microfluídica consigue rendimientos similares a las selecciones biológicas evitando la complejidad de las mismas. En el marco del proyecto MetaFluidics, que coordinamos desde este grupo, hemos implementado una plataforma de “sorting” basada en fluorescencia para encontrar hidrolasas termoestables en ambientes termales y otras enzimas de relevancia para biocatálisis. Para ello, ha habido que desarrollar instrumentación a la vez que nuevos huéspedes y protocolos para expresión funcional compatible con ensayos enzimáticos a altas temperaturas.
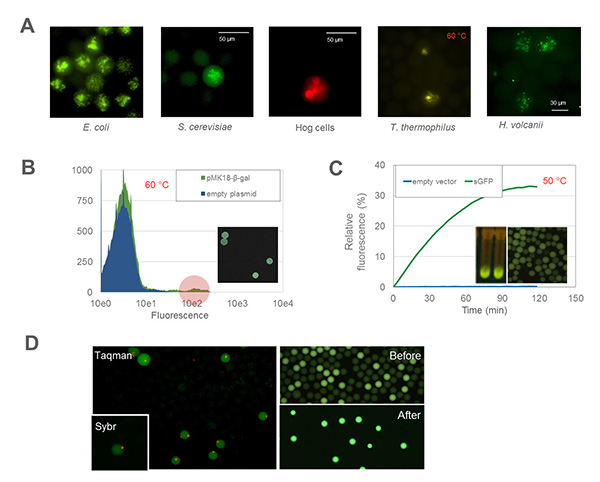
Uso de microgotas en cribados de ultra-alta capacidad. A) Crecimiento de microorganismos y células de mamíferos. B) Cribado de termozimas recombinantes expresadas en T. thermophilus. C) Expresión in vitro de termozimas utilizando la transcripción/traducción in vitro derivada de T. thermophilus. D) PCR digital para detectar microorganismos raros (materia biológica oscura) y genes. Crédito de las figuras: M. Almendros, A. Lopes y M. Sánchez.

| Apellidos | Nombre | Laboratorio | Ext.* | Categoría profesional | |
|---|---|---|---|---|---|
| Blas Muñoz | Laura | 108 | 4525 | laura.blas(at)cbm.csic.es | Titulado Sup.de Actividades Técn. y Profes. GP1 |
| Bosch Reñe | Sandra | 108 | 4525 | sbosch(at)cbm.csic.es | Titulado Superior Grado de Doctor |
| Cecchini | Davide Agostino | 108 | 4525 | davide.cecchini(at)uam.es | Investigador Doctor |
| Hidalgo Huertas | Aurelio | 108 | 4527 | ahidalgo(at)cbm.csic.es | Profesor Titular Universidad, GA |
| Lorente Arévalo | Álvaro | 108 | 4525 | alorente(at)cbm.csic.es | Titulado Sup.de Actividades Técn. y Profes. GP1 |
| Margot | Léa | 108 | 4524 | Titulado Sup.de Actividades Técn. y Profes. GP1 | |
| Martínez Salvador | John | 108/127 | 4525/4569 | john.martinez(at)cbm.csic.es | Tco. de Investigación y Laboratorio |
| Nejda Hamdoun | Nargisse | 108 | 4524 | n.nejda(at)cbm.csic.es | Titulado Sup.de Actividades Técn. y Profes. GP1 |
| Ortega Plaza | Carmen | 108 | 4525 | cortega(at)cbm.csic.es | Tco. de Investigación y Laboratorio |
| Palacios Azcona | Aitor | 108 | 4525 | Estudiante TFG | |
| Pérez Arnaiz | Patricia | 108 | 4498 | pparnaiz(at)cbm.csic.es | Profesor Ayudante Doctor Universidad |
| Saiz Alvarez | Laura Pilar | 108 | 4525 | Titulado Sup.de Actividades Técn. y Profes. GP1 |
Publicaciones relevantes:
- Mate DM, Rivera NR, Sanchez-Freire E, Ayala JA, Berenguer J and Hidalgo A (2019) Thermostability enhancement of the Pseudomonas fluorescens esterase I by in vivo folding selection in Thermus thermophilus. Biotech. Bioeng. doi: 10.1002/bit.27170
- Verdú C, Sanchez-Freire E, Ortega C, Hidalgo A, Berenguer J* and Mencía M* (2019) A modular vector toolkit with a tailored set of thermosensors to regulate gene expression in Thermus thermophilus. ACS Omega doi: 10.1021/acsomega.9b02107
- Castillo J, Caminata Landriel S, Sánchez Costa M, Taboga OA, Berenguer J, Hidalgo A, Ferrarotti SA, Costa H (2018) A single mutation in cyclodextrin glycosyltransferase from Paenibacillus barengoltzii changes cyclodextrin and maltooligosaccharides production. Protein Eng. Des. Sel. 31: 399-407. doi. 10.1093/protein/gzy034
- Consolati T, Bolívar JM, Petrasek Z, Berenguer J, Hidalgo A, Guisán JM, and Nidetzky B (2018) Bio-based pH internally sensitive materials: immobilized yellow fluorescent protein as optical sensor for spatiotemporal mapping of pH inside porous matrices. ACS Appl. Mater. Inter. 10: 6858-6868. doi: 10.1021/acsami.7b16639
- Ribeiro AL, Mencía M and Hidalgo A (2018) A brief guide to the high-throughput expression of directed evolution libraries. Methods Mol. Biol. 1685: 131-143. doi: 10.1007/978-1-4939-7366-8_7
- Ribeiro AL, Sánchez M, Hidalgo A, and Berenguer J (2017) Stabilization of enzymes by using thermophiles. Methods Mol. Biol. 1465: 297-312. doi: 10.1007/978-1-4939-7183-1_21
- Berenguer J, Mencía M, and Hidalgo A (2017) Are in vivo selections on the path to extinction? Microb. Biotechnol. 10: 46-49. doi: 10.1111/1751-7915.12490
- Vettone A, Serpe M, Hidalgo A, Berenguer J, Del Monaco G, Valenti A, Rossi M, Ciaramella M and Perugino G (2016) A novel thermostable protein-tag: optimization of the Sulfolobus solfataricus DNA-alkyl-transferase by protein engineering. Extremophiles 2016, 20: 1-13. Doi: 10.1007/s00792-015-0791-9
- Hidalgo A, Schließmann A and Bornscheuer UT (2014) One-pot Simple methodology for CAssette Randomization and Recombination for focused directed evolution (OSCARR). Methods. Mol. Biol. 1179: 207-212. doi: 10.1007/978-1-4939-1053-3_14
- Hidalgo A and Berenguer J (2013) Biotechnological applications of Thermus thermophilus as host. Curr. Biotechnol. 2: 304-312. doi: 10.2174/18722083113076660030
- Torres LL, Cantero A, Del Valle M, Marina A, Lopez-Gallego F, Guisan JM, Berenguer J and Hidalgo A (2013) Engineering the substrate specificity of a thermophilic penicillin acylase from Thermus thermophilus. Appl. Environ. Microbiol. 79: 1555-1562. doi: 10.1128/AEM.03215-12
- Torres LL, Ferreras ER, Cantero A, Hidalgo A and Berenguer J (2012) Functional expression of a penicillin acylase from the extreme thermophile Thermus thermophilus HB27 in Escherichia coli. Microb. Cell Fact. 11: 105. doi: 10.1186/1475-2859-11-105.
- Torres LL, Schließmann A, Schmidt M, Silva-Martin N, Hermoso JA, Berenguer J, Bornscheuer UT and Hidalgo A (2012) Promiscuous enantioselective (-)-γ-lactamase activity in the Pseudomonas fluorescens esterase I. Org. Biomol. Chem. 10: 3388-3392. doi:
10.1039/C2OB06887G.
Tesis dirigidas:
- Yamal Al-ramahi González (2013) Ingeniería de proteínas fluorescentes y aplicaciones de localización celular en microorganismos termófilos. Universidad Autónoma de Madrid. Directores: Aurelio Hidalgo y José Berenguer.
- Noé R. Rivera (2013) Termoestabilización de proteínas de interés biológico. Universidad Autónoma de Madrid. Directores: Aurelio Hidalgo y José Berenguer.
Tesis en curso:
- Mercedes Sánchez Costa. Descubrimiento y caracterización de enzimas termófilas con implicación en biocatálisis. Directores: Aurelio Hidalgo y José Berenguer.
- Sandra Bosch Reñé. Selección de biocatalizadores termoestables para química verde empleando bioherramientas basadas en microorganismos termófilos Director: Aurelio Hidalgo.
- Jorge Bravo Villanueva. Desarrollo de procesos de cribado masivo basados en bioherramientas de organismos termófilos. Director: Aurelio Hidalgo.
Financiación:
- Desarrollo de sistemas para la selección de proteínas termoestables mediante expresión in vivo e in vitro a alta temperatura. Entidad financiadora: UAM. UAM/89. Período: 01/02/2017-31/12/2021. Tipo de participación: IP.
- Advanced toolbox for rapid and cost-effective functional metagenomic screening: microbiology meets microfluidics (METAFLUIDICS). Entidad financiadora: Unión Europea H2020. GA 685474. Período: 01/06/2016-30/05/2020. Tipo de participación: Coordinación.
- Sustainable industrial processes based on a C-C bond-forming enzyme platform (CarbaZymes). Entidad financiadora: Unión Europea H2020. GA 635595. Período: 01/04/2015-31/03/2019. Tipo de participación: Socio.
- Ultrahigh-throughput platform for the screening of thermostable proteins by thermophilic in vitro transcription-translation and microfluidics (HOTDROPS). Entidad financiadora: Unión Europea FP7. GA 324439. Período: 01/06/2013-30/05/2017. Tipo de participación: Socio.
- Selección de alta eficiencia de biocatalizadores termoestables para química verde empleando microorganismos termófilos modificados. Entidad financiadora: Ministerio de Economía y Competitividad. Período: 01/01/2013-31/12/2016. Tipo de participación: Co-IP.




















